Capitolo 5 Analisi statistica di due variabili (bivariata)
Prima di procedere all’analisi di due variabili, considerando i diversi casi possibili, 2 variabili entrambe qualitative, 1 variabile quantitativa e 1 qualitativa e, infine, 2 variabili quantitative, conviene introdurre alcuni strumenti che possono esser utili per confrontare distribuzioni di dati. Si considerano, in particolare, strumenti grafici che permettono di confrontare dati empirici con dati conformi a un modello teorico oppure di tecniche per confrontare due distribuzioni empiriche in due o più insiemi di dati osservati. Alcuni di tali strumenti saranno poi utili in seguito anche per le tecniche di analisi bivariata.
5.1 Confrontare distribuzioni
5.1.1 Confronto fra distribuzioni empiriche e teoriche
In particolare per l’analisi di variabili quantitative, risulta spesso utile poter disporre di strumenti per verificare se i dati osservati si conformano a qualche modello teorico. Ci si potrebbe chiedere se i dati abbiano una distribuzione che si discosta poco da un modello distributivo noto e del quale sono note le proprietà come potrebbe essere, ad esempio, la gaussiana.
Il problema può essere affrontato con un approccio formale e con tecniche di inferenza statistica e/o in modo più informale con strumenti grafici. Spesso in realtà i due approcci sono usati in forma complementare. Si rinvia a testi di statistica inferenziale per un approccio più formale al problema, gli strumenti grafici adatti a tale scopo sfrutteranno essenzialmente alcune delle tecniche di visulaizzazione già introdotte per l’analisi di variabili quantitative.
5.1.1.1 Confronto fra funzione di ripartizione empirica e teorica
La funzione di ripartizione empirica è stata già introdotta nel precedente capitolo. Uno strumento immediato per confrontare la distribuzione dei dati osservata si ottiene sovrapponendo alla funzione di ripartizione empirica dei dati la funzione di ripartizione della variabile che si vuole confrontare. Tale funzione di ripartizione deve quindi essere completamente nota.
Per illustrare tale grafico in R si mettono a confronto i dati empirici con quelli costituiti da valori pseudocasuali generati da una variabile aleatoria nota. Ricordiamo che uno strumento importante di R sono le funzioni speciali per trattare modelli aleatori per i quali sono stati già mostrati alcuni esempi.
Si tratta di funzioni che consentono di valutare alcune quantità rilevanti per specifiche variabili casuali appartenenti alle principali famiglie parametriche. Si può ottenere, ad esempio, molto facilmente la funzione di probabilità (o di densità), la funzione di ripartizione, la funzione quantile e si possono generare numeri casuali per distribuzioni di probabilità, come la Binomiale, la Poisson, la Normale, la Gamma e molte altre.
Le funzioni contengono un nome che identifica la famiglia di variabili casuali e se tale nome è preceduto da d viene calcolata la probabilità (o la densità), se preceduto da p si ottiene la funzione di ripartizione, con q si ottiene la funzione dei quantili e se preceduta da r si generano valori casuali.
Utilizzando l’help in linea di R (?Distributions) si ottiene l’elenco tutte le distribuzioni di probabilità implementate nell abase di R. Ad esempio, la funzione pnorm(x,m,s) valuta la funzione di ripartizione in \(x\) per un modello gaussiano con media \(m\) e deviazione standard \(s\).
Molte altre famiglie di variabili aleatorie sono disponibili in pacchetti dedicati all’analisi di dati per specifici ambiti applicativi. Si simulano ora dei dati da una gaussiana e si ottiene il grafico della funzione di ripartizione empirica. Ci si propone di confrontare la funzione di ripartizione empirica dei dati osservati con una Gaussiana di media e varianza nota.
# si generano i dati pseudocasuali da una gaussiana standard
set.seed(1111)
dat1<-rnorm(50)
plot(ecdf(dat1),cex=.5,
main="confronto ripartizione empirica e teorica gaussiana")
curve(pnorm(x), add=TRUE)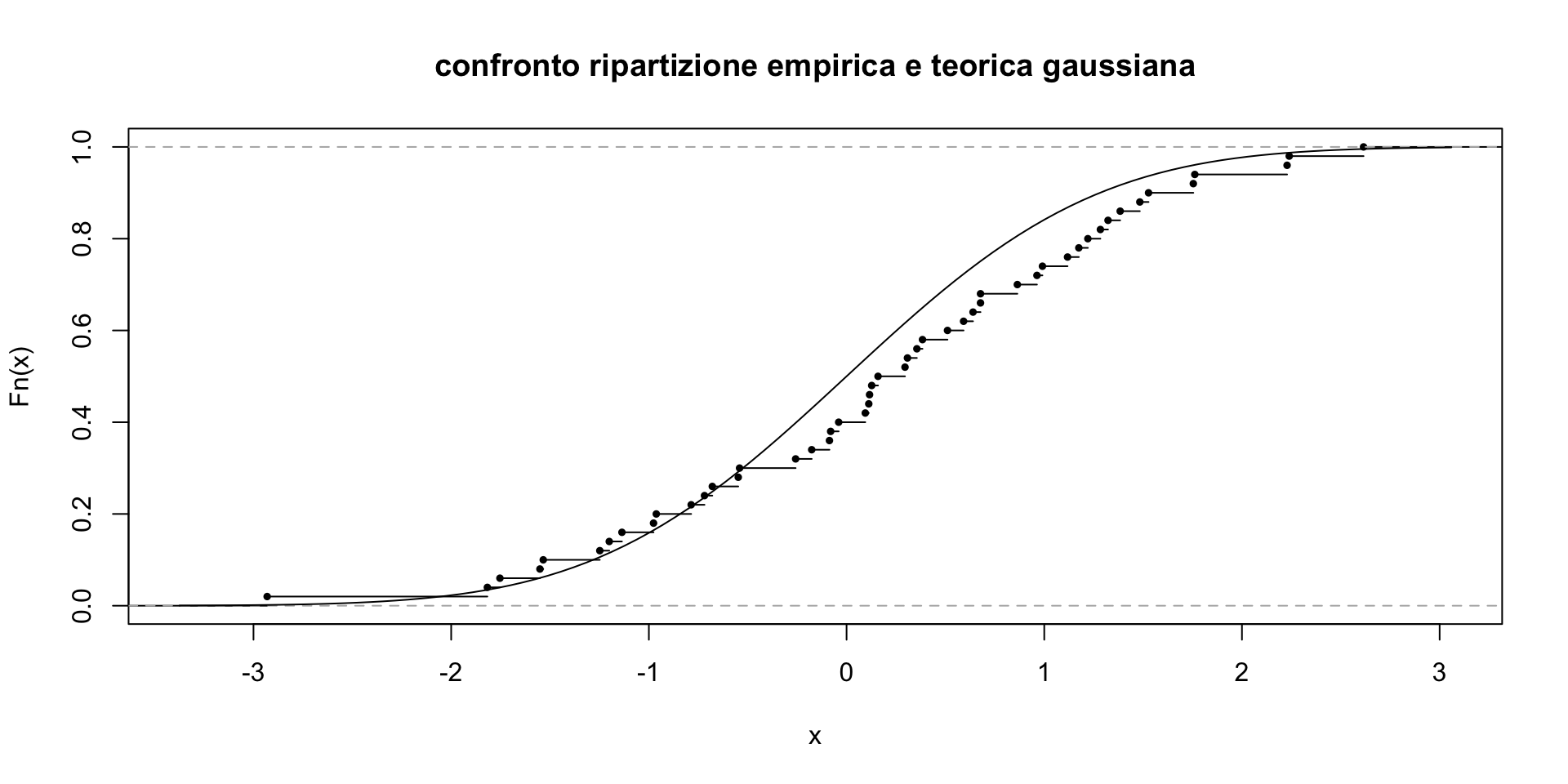
# si prova ora a generare da una distribuzione t di student con 1
# grado di libertà (simile alla gaussiana ma con code più pesanti)
# e si confrontno i due insiemi di dati con una gaussiana
# di media e varianza nulla
dat2<-rt(50,2)
plot(ecdf(dat2), cex=.5, verticals=TRUE,
main="confronto ripartizione empirica dati tratti da t
con teorica gaussiana")
curve(pnorm(x), add=TRUE)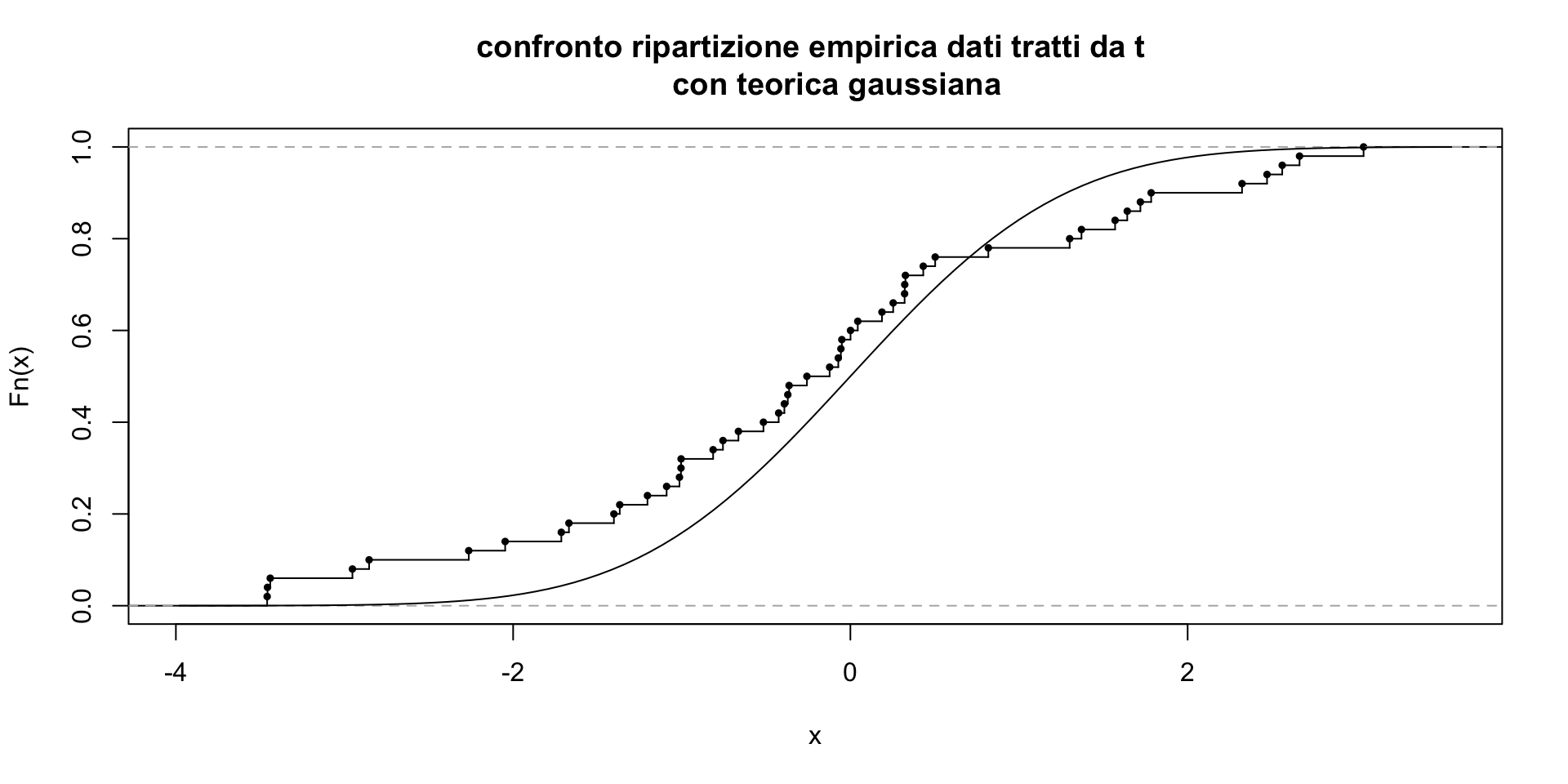
Come si vede, nel secondo caso c’è uno scostamento sensibile fra la funzione di ripartizione empirica per i dati generati da una distribuzione con code pesanti e una ditribuzione gaussiana. In effetti, buona parte delle procedure statistiche per verificare se i dati osservati sono conformi a una distribuzione teorica si basano sul confronto fra tali curve.
In particolare la distanza fra le due curve viene spesso riassunta dalla statistica di Kolmogorov-Smirnov \(D\) definita come \[ D=\underset{x}{sup}|\hat{F}(x)-F(x)|\] cioè la massima distanza verticale fra le due curve.
Si noti che il confronto può essere fatto anche con una distribuzione teorica per la quale si ipotizza la conoscenza della famiglia ma non la conoscenza dei parametri. Ad esempio, si potrebbe ipotizzare i dati provengano da una gaussiana per la quale media e varianza sono ignote. In tal caso si possono utilizzare lea media e la varianza calcolate sui dai dati raccolti e usarle come fossero i valori veri. Il confronto grafico fra le curve resta uno strumento utile anche in questo caso ma le proprietà statistiche, ad esempio quelle della statistica \(D\) non sono le stesse e questo ha conseguenze nell’ambito dei metodi statistici inferenziali.
5.1.1.2 Confronto delle funzioni di densità (empirica e teorica)
Un altro modo per confrontare una variabile osservata con con un modello teorico è quello di confrontare la funzione di densità del modello teorico (che si suppone sempre completamente noto). Si può quindi sovrapporre la funzione di densità teorica alle sue versioni empiriche tratte dai dati. Ad esempio, sovrapporre la funzione di densità teorica all’istogramma (che ricordiamo essere une versione grezza ed emprica della funzione di densità)
par(mfrow=c(1,2))
dat3<-rnorm(100) # si generano dati da una gaussiana standard
hist(dat3,prob=T, xlim=c(-5,5), ylim=c(0,0.6))
# e si sovrappone la funzione di densità della gaussiana standard
curve(dnorm(x,0,1), col=2, lwd=2, add=TRUE)
# Ora si può provare con una numerosità dei dati più elevata
dat32<-rnorm(1000) # generiamo dati da una gaussiana standard
hist(dat32,prob=T, xlim=c(-5,5), ylim=c(0,0.6))
# sovrapponiamo la funzione di densità della gaussiana standard
curve(dnorm(x,0,1), col=2, lwd=2, add=TRUE)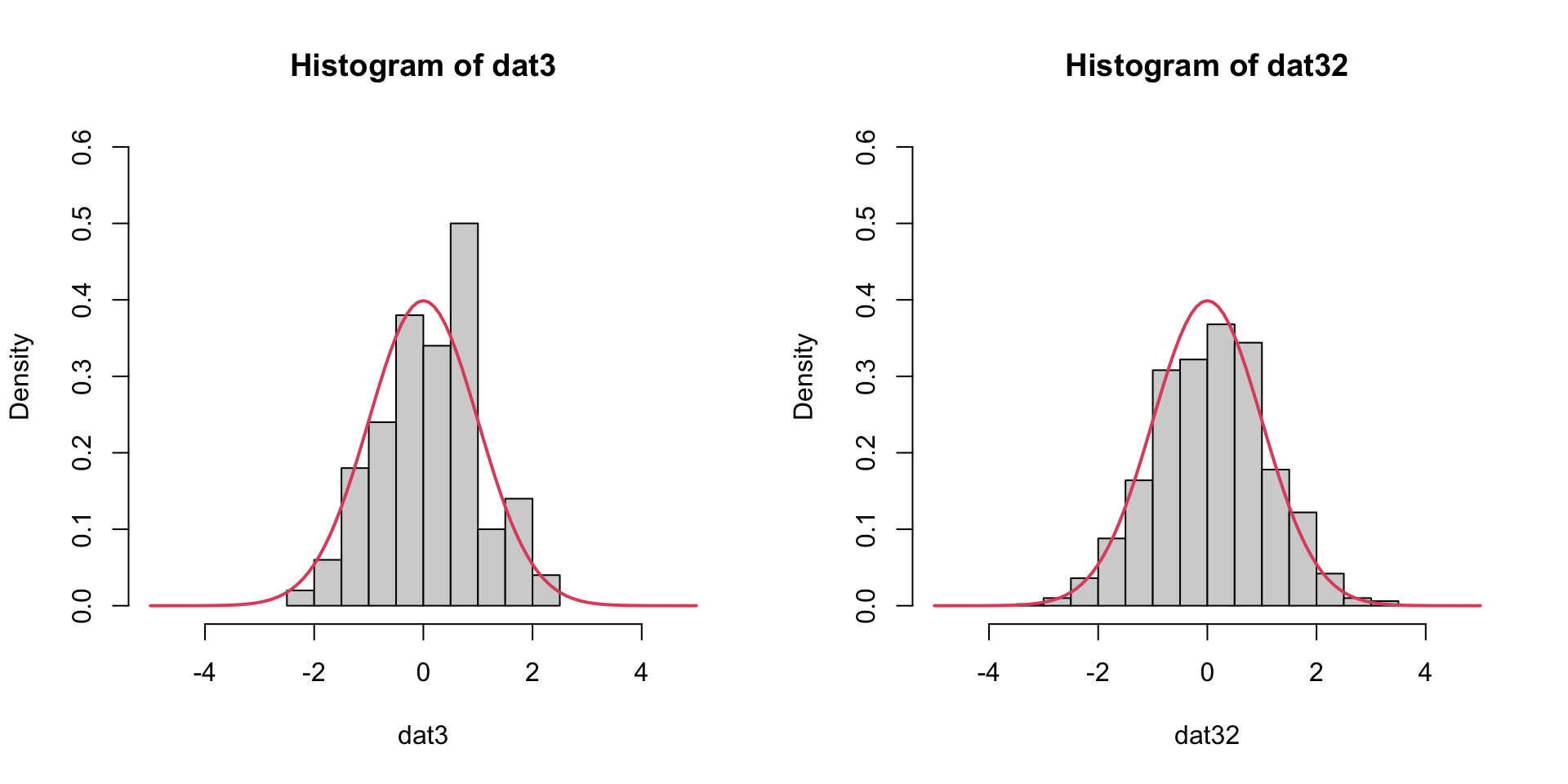
Con 100 dati, come si vede, l’istogramma risulta instabile e la somiglianza è vaga; come regola generale non vanno sopravvalutate le differenze nei grafici se si dispone di pochi dati. Con numerosità maggiori (e quindi con più classi) la somiglianza fra istogramma e modello teorico (nel caso in cui i dati peraltro provengono da quel modello teorico) aumenta.
In alternativa, si potrebbe usare la curva di densità empirica ottenuta con il metodo del nucleo. Si può provare a vedere se i dati empirici, generati da una distribuzione con code pesanti, come la t di student con 1 grado di libertà, differiscono dal modello teorico gaussiano.
dat4<-rt(1000,1) # si generano i dati da una t di studente con 1 gdl
plot(density(dat4), xlim=c(-6,6), ylim=c(0,0.6))
# se si sovrappone la funzione di densità di una gaussiana,
# dovremmo notare differenze sulle code
curve(dnorm(x,0,1), col=2, lwd=2, add=TRUE)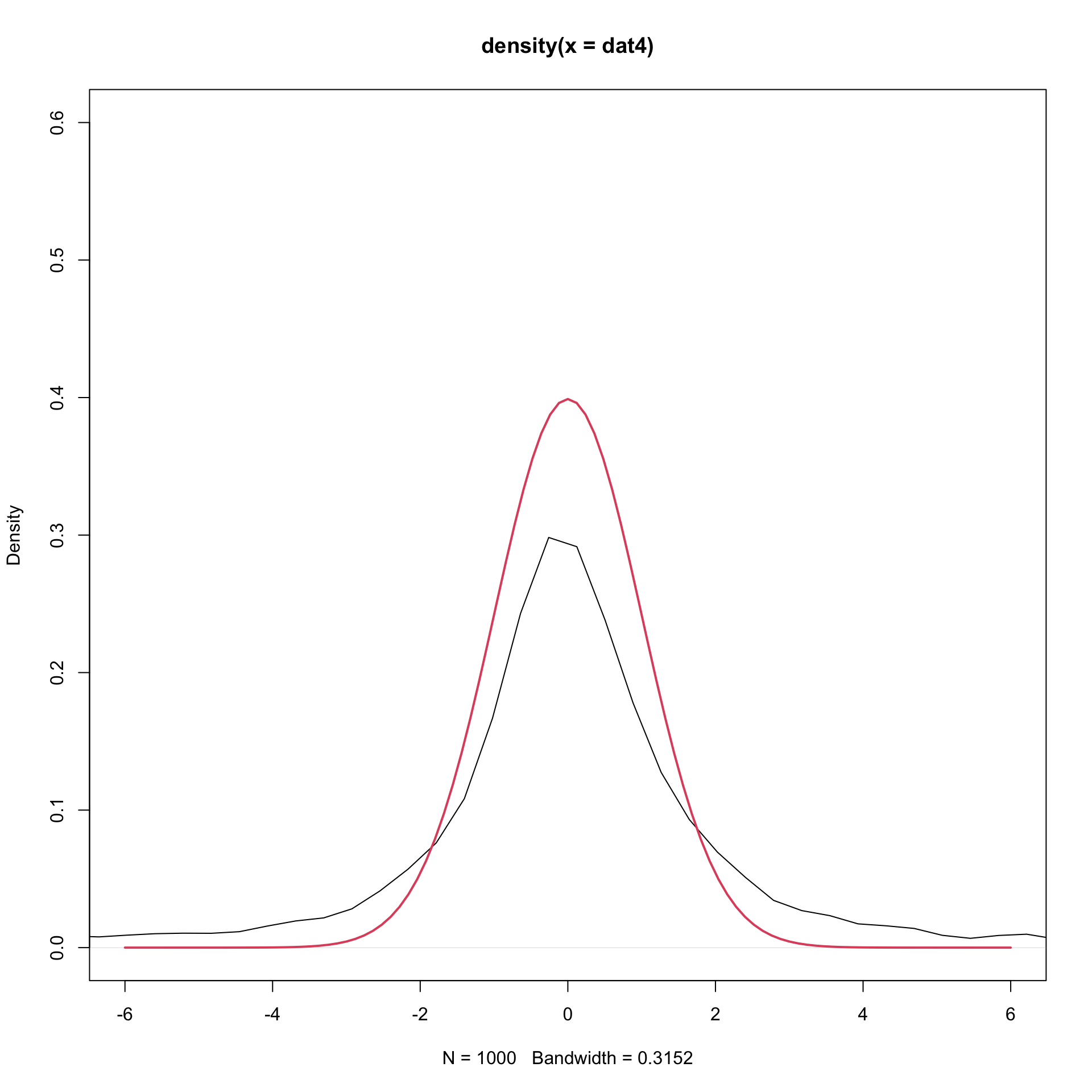
5.1.1.3 Confronto dei quantili: il grafico quantile-quantile
Per una variabile aleatoria continua \(X\) è definita la funzione dei quantili \(F^{-1}(p)=x_p\) che è la funzione inversa della funzione di ripartizione. Essa associa ad ogni valore \(p \in [0,1]\) il corrispondente quantile \(x_p\).
Si noti che in R per i diversi modelli teorici si ottiene il quantile di una variabile aleatoria appartenente a una famiglia parametrica premettendo q al nome che identifica la famiglia. Quindi qnorm(p,m,s) fornisce per un dato \(p\) il quantile della famiglia gaussiana di parametri dati.
Per un insieme di dati quantitativi osservati è possibile ottenere il grafico dei quantili empirici. Si ricorda che il valore \(x_{(i)}\) rappresenta il quantile empirico \(x_{\frac{i}{n}}\). Nel seguito usiamo la convenzione per cui il valore $x_{(i)}rappresenta il quantile \(x_{\frac{i-0.5}{n}}\)
set.seed(2222)
dat5<-rnorm(100) # si generano i dati da una gaussiana standard e si
# ottengono i quantili empirici
dat5s<-sort(dat5)
qe<-((1:100)-0.5)/100
# e poi si traccia il grafico dei quantili
plot(qe, dat5s)
# cui si sovrappone il grafico dei quantili di una gaussiana standard
curve(qnorm(x,0,1), lwd=2, add=TRUE)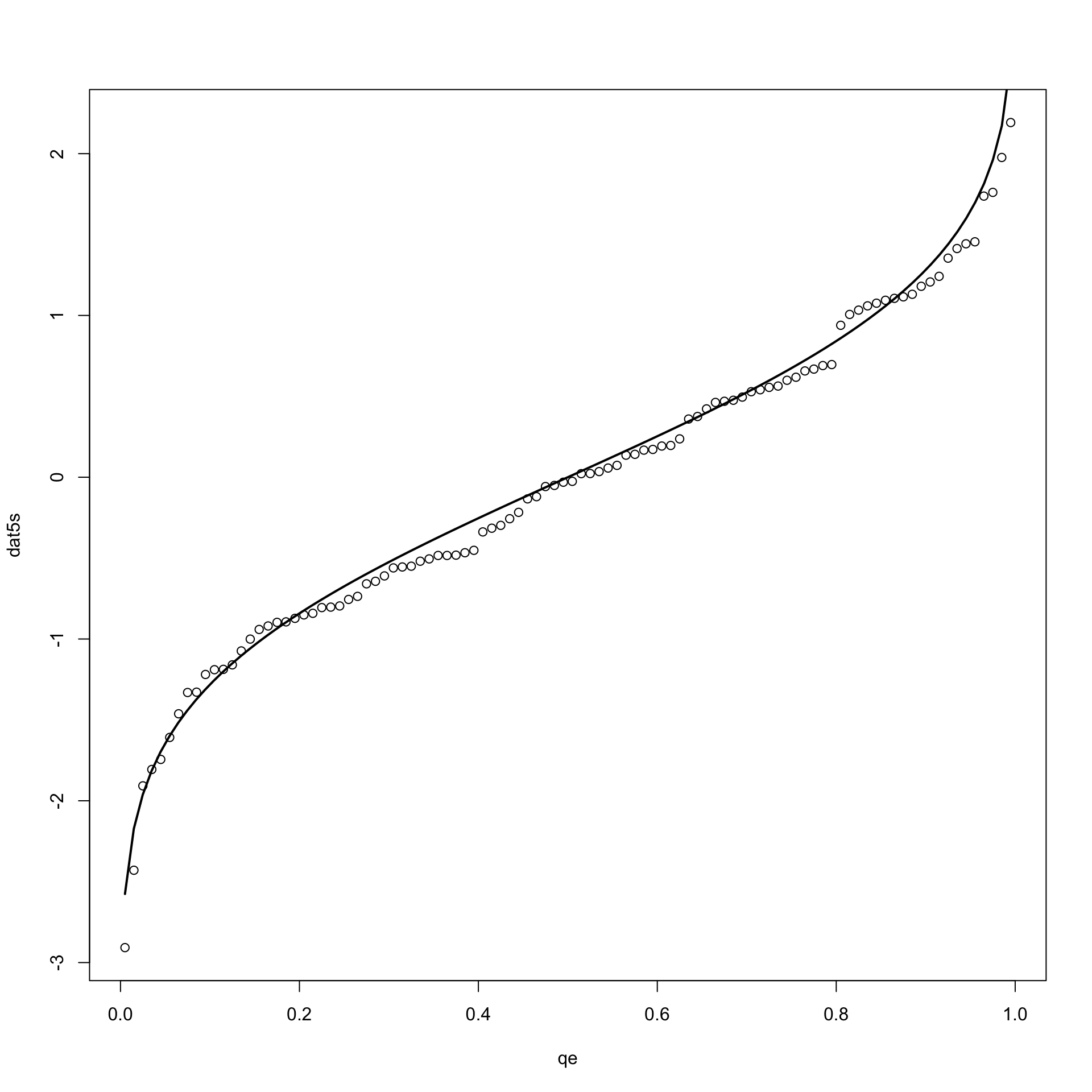
Come si vede le due curve sono molto vicine a indicare che i dati provengono da quel modello teorico.
Si può tuttavia pensare a una strategia di confronto dei quantili diversa. Ci aspettiamo che un quantile empirico sia molto vicino a quello teorico se le distribuzioni si somigliano. Se quindi si confrontano i quantili teorici \(x_p\) con i quantili empirici ottenuti per ogni valore \(p=\frac{i}{n}\) in corrisponedenza di ogni dato osservato, essi dovrebbero disporsi lungo la retta bisettrice del I e IV quadrante se i dati provengono dal medesimo modello. Posso quindi considerare tutti i valori \(p=\frac{i-0.5}{n}\) e a ciascun valore \(x_{(i)}\) associare il quantile teorico.
Questa ultima curva rappresenta un importantissimo strumento per valutare l’aderenza fra dati empirici e modello teorico noto col nome di grafico quantile-quantile abbreviato spesso in qqplot.
Non è quindi sorprendente che esista una funzione per tracciare questo plot. In particolare per il confronto con una gaussiana si può usare la funzione qqnorm() (questo è un caso particolare della funzione qqplot() che illustreremo più avanti).
par(mfrow=c(1,2))
# prima si ottiene il grafico usando la definizione e la funzione plot()
plot(qnorm(qe), dat5s, main="grafico quantile-quantile per confronto con gaussiana")
abline(0,1)
# Ora si utilizza direttamente la funzione qqnorm()
qqnorm(dat5s, main="grafico qqnorm per confronto con gaussiana")
qqline(dat5s) # aggiunge la linea per verificare il confronto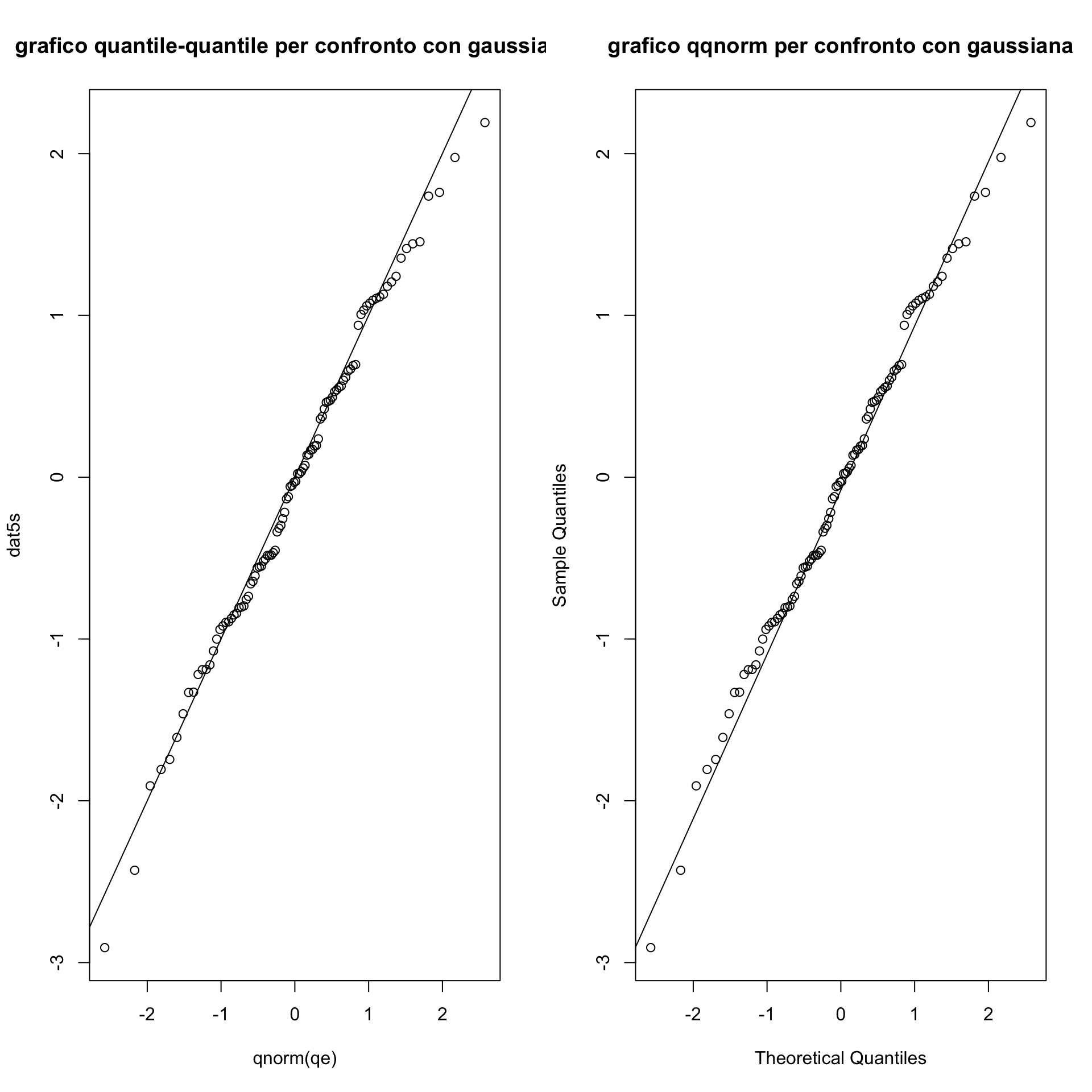
Proviamo ora a vedere cosa accade se confrontiamo i quantili calcolati per i dati tratti da un’altra distribuzione (la solita t di student con 1 gdl) con la gaussiana standard.
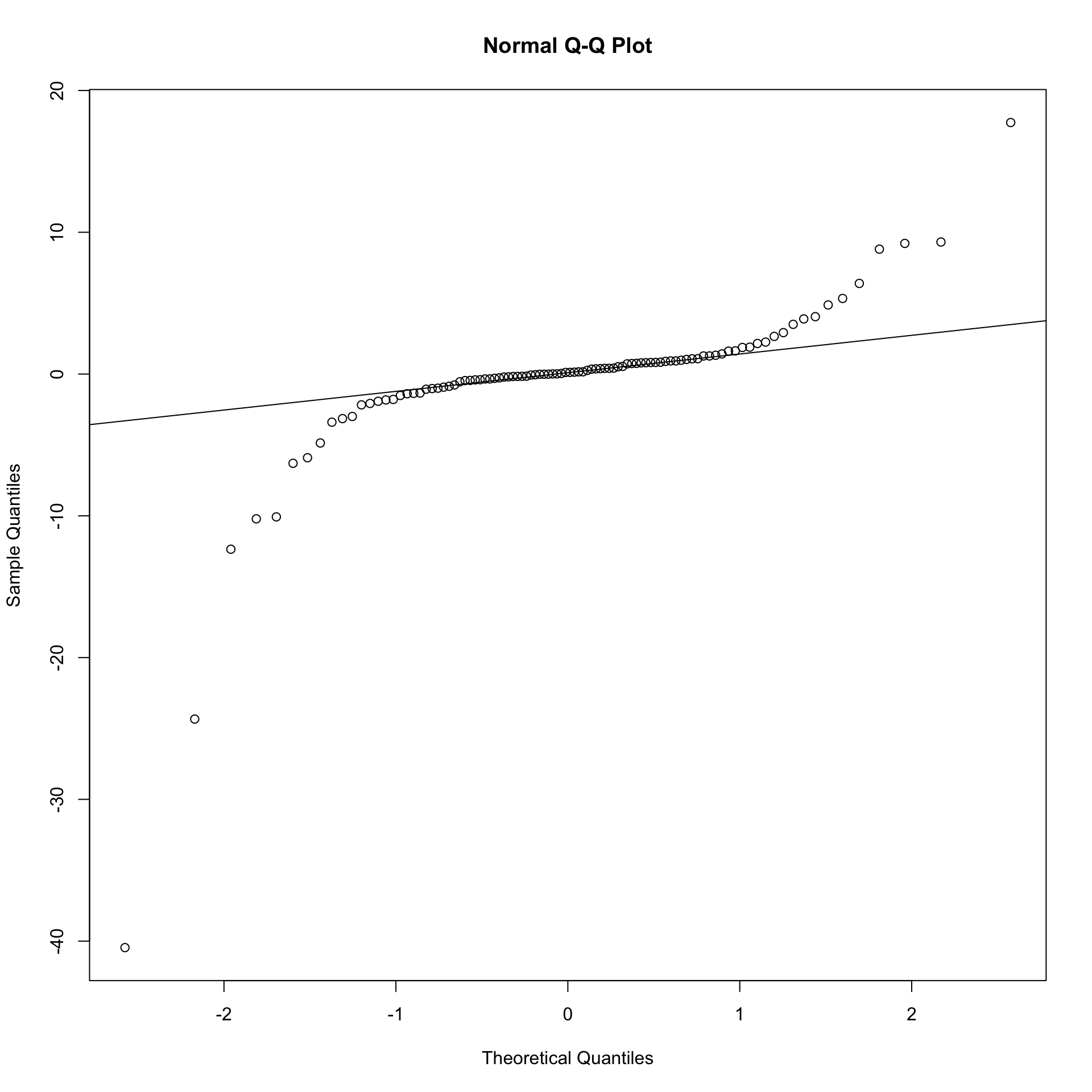
5.1.2 Strumenti grafici per il confronto fra due o più insiemi di dati osservati
I grafici introdotti per le variabili statistiche (in particolare quelle quantitative) e alcuni strumenti illustrati per il confronto tra distribuzione empirica e teorica possono essere utilizzati per confrontare due (o anche più di due) insiemi di dati osservati.
Si tratta in questo caso di affiancare o sovrapporre le rappresentazioni grafiche già introdotte.
5.1.2.1 Box-plot affiancati
Come già discusso, il diagramma a scatola con baffi è una rappresentazione molto usata per rappresentare sinteticamente la distribuzione di un insieme di dati quantitativi. E si è anche osservato come essa possa essere efficace per rappresentare anche piccoli insiemi di dati. In questo ultimo caso, ad esempio, usare un istogramma (con il conteggio dei casi entro classi di valori) non è sempre una buona idea.
Se si dispone di due insiemi di dati per i quali si osserva la medesima variabile si possono affiancare (o sovrapporre) i relativi boxplot.
I dati che seguono (piuttosto famosi perchè introdotti da Ronald Fisher nel 1936 e spesso usati per illustrare alcune tecniche di analisi dei dati) riportano insiemi di misure relative a diverse specie di fiori di iris.
data(iris)
boxplot(iris$Sepal.Length[iris$Species=="virginica"],
iris$Sepal.Length[iris$Species=="versicolor"],
names=(c("","versicolor")))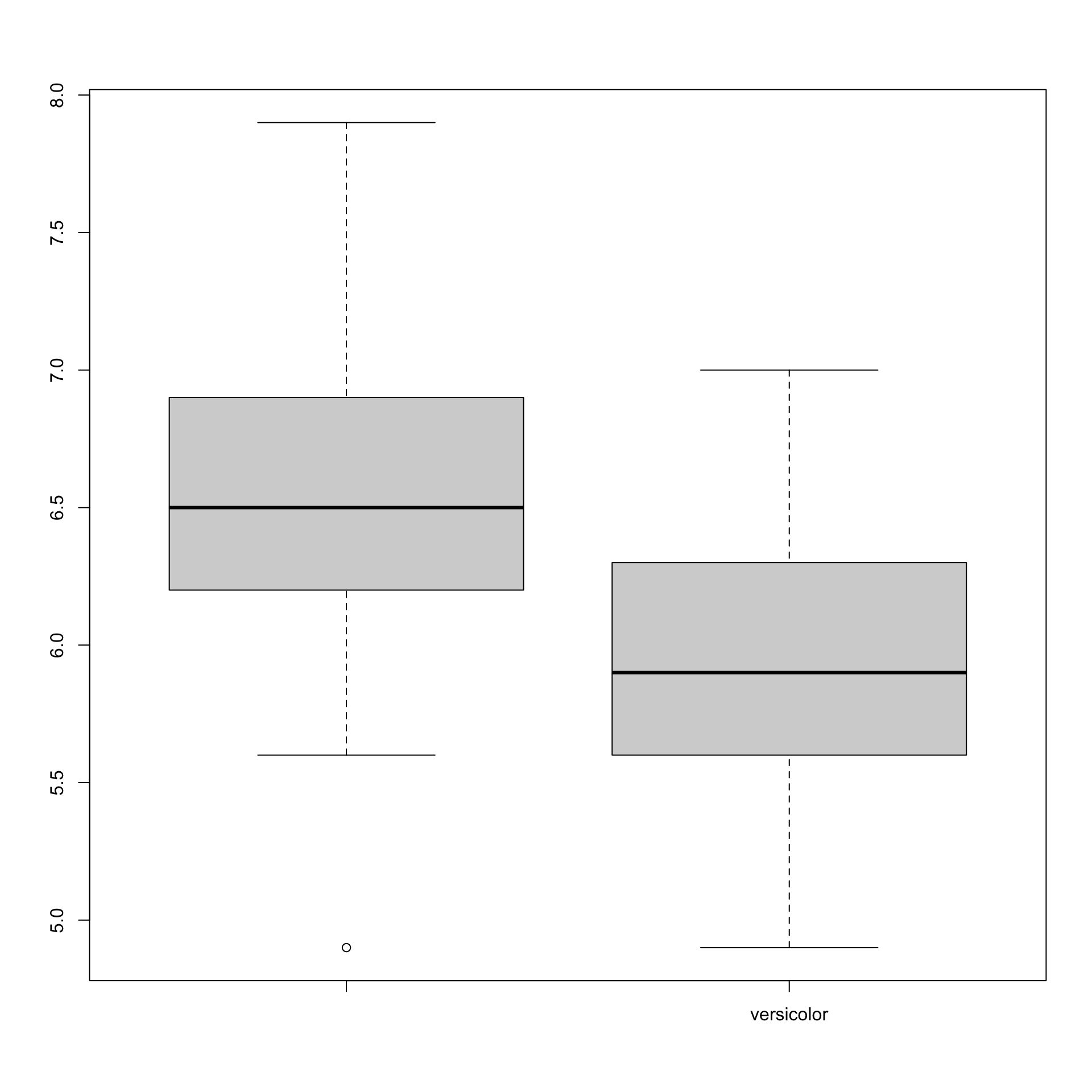
In realtà, per i boxplot è molto semplice ottenere una versione in cui si affiancano le rappresentazioni delle distribuzioni di una variabile quantitativa anche più di due gruppi.
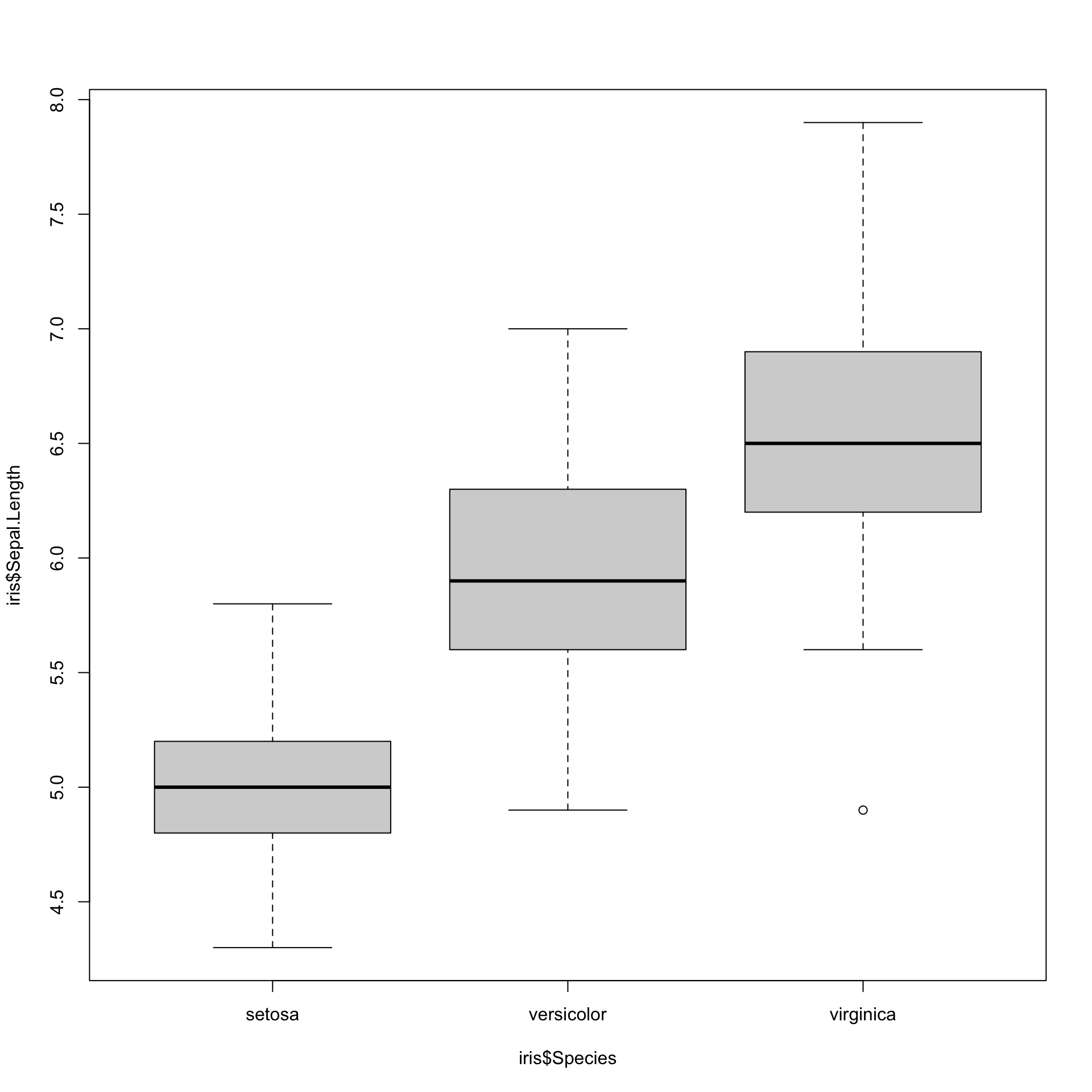
# si vedrà che il simbolo "~" è importante per definire quella che chiameremo
# in R "formula", in cui una variabile viene studiata condizionatamente
# a un'altra variabile (in questo caso a un fattore).
# Si noti che si potrebbe pure scrivere
# boxplot(Sepal.Length~Species, data=iris)
# ottenendo il medesimo risultato senza premettere a ciascuna
# variabile il nome del dataframe che le contiene.5.1.2.2 Confronto tra le funzioni di ripartizione empiriche
È facile ottenere per i diversi gruppi da confrontare le rispettive funzioni di ripartizione empiriche e sovrapporle nel grafico. Si vedrà ancora un esempio con i dati iris.
Anche in questo caso possiamo confrontare le funzioni di ripartizione empirica dei due gruppi.
plot(ecdf(iris$Sepal.Length[iris$Species=="setosa"]), verticals=TRUE, xlim=c(4,7.5),
main="confronto fra funzioni di ripartizioni empiriche di due gruppi")
plot(ecdf(iris$Sepal.Length[iris$Species=="versicolor"]),
verticals=TRUE, col=2, add=TRUE)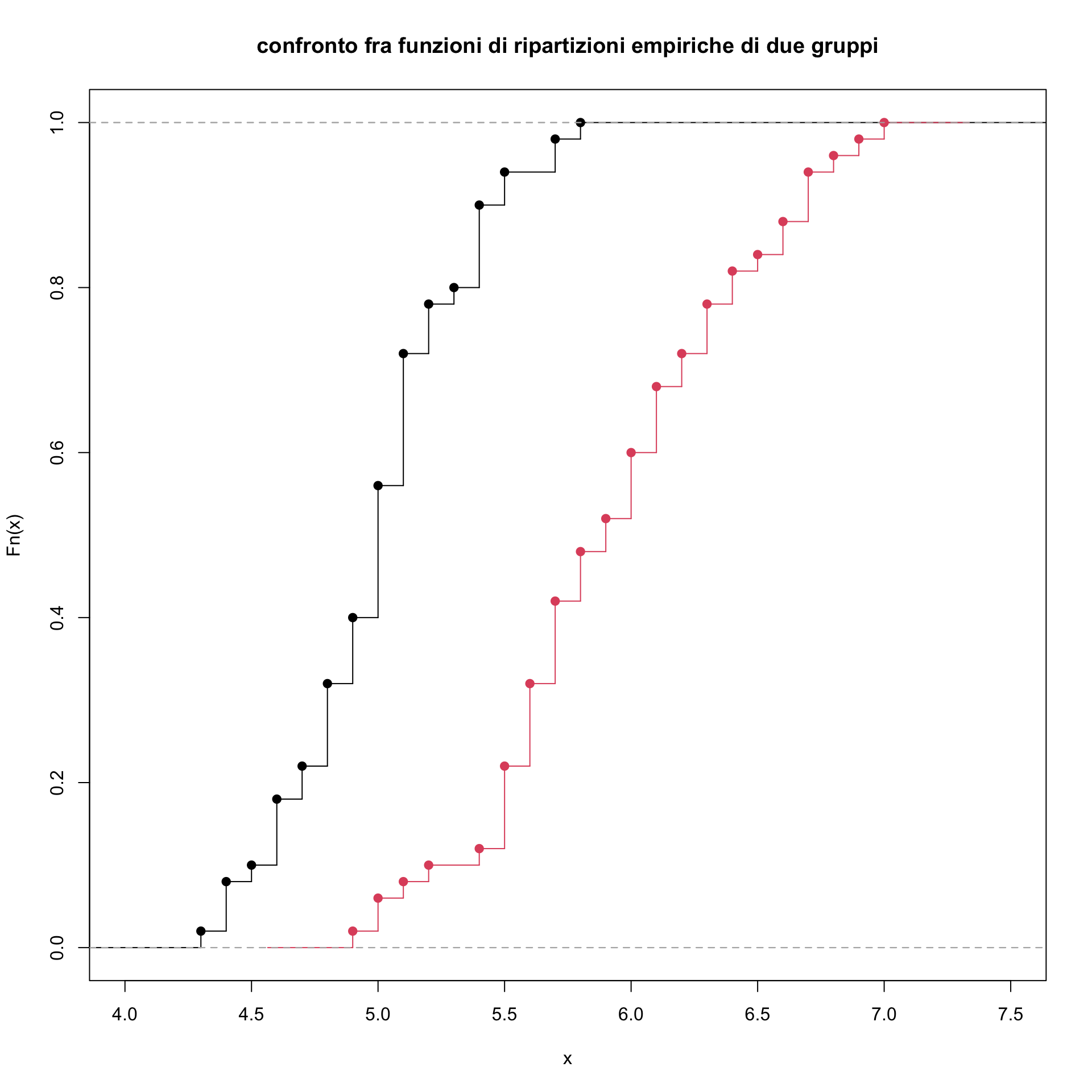
Come si vede la funzione di ripartizione empirica ricavata dai dati per la specie versicolor \(X_v\) è spostata a destra rispetto a quella della specie setosa \(X_s\) (cioè \(\hat{F}_v(x)) \leq \hat{F}_s(x_s))\).
Ovviamente si potrebbe aggiungere anche quella di un altro gruppo al grafico con il rischio che però risulti meno chiaro.
Anche in questo caso si potrebbe usare la statistica di Kolmogorof-Smirnof per misurare la distanza fra le due curve definita, ancora una volta, come il massimo scostamento fra le due curve. \[ D=\underset{x}{sup}|\hat{F}_s(x)-\hat{F}_v(x)|\]
5.1.2.3 Confronto tra le funzioni di densità empiriche
Si potrebbero anche confrontare le funzioni di densità empirica dei due gruppi. In particolare, si noti notare come sovrapporre istogrammi non conduca a una rappresentazione grafica efficace per mettere in luce le differenze fra i due gruppi.
# per brevità di notazione
hist(iris$Sepal.Length[iris$Species=="versicolor"], xlim=c(4,8.5), freq=FALSE,
, xlab="lunghezza sepalo", main="sovrapposizione di istogrammi di due gruppi")
hist(iris$Sepal.Length[iris$Species=="virginica"], border=2, freq=FALSE,
density=4.5, add=TRUE)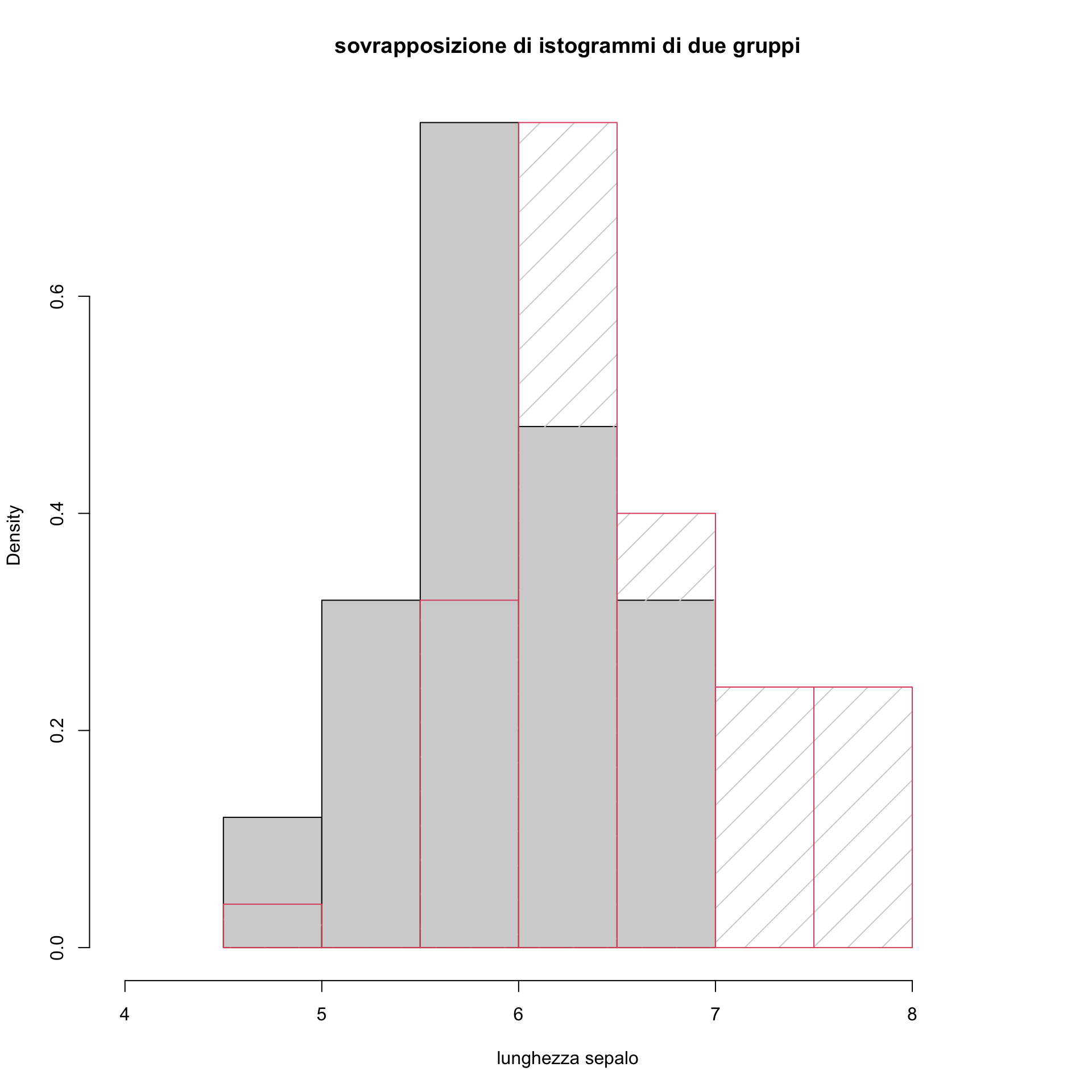
L’istogramma ricavato dai dati per la specie virginica \(X_v\) è spostata a destra rispetto a quella della specie versicolor come si vedeva benissimo con i box plot. Non è però molto efficace la rappresentazione grafica
Più chiaro risulta il grafico che sovrappone le curve di densità con il metodo del nucleo.
# per brevità di notazione si ottengano dei vettori con i
# dati sulle lunghezze dei sepali per i tre gruppi
virgi<-iris$Sepal.Length[iris$Species=="virginica"]
versi<-iris$Sepal.Length[iris$Species=="versicolor"]
setosa<-iris$Sepal.Length[iris$Species=="setosa"]
plot(density(virgi), xlim=c(4,8.5), xlab="lunghezza sepalo",
main="sovrapposizione di densità col metodo del nucleo")
lines(density(versi), col=2)
legend(x=7.5,y=0.6,legend=c("versicolor","virginica"), fill=c(2,1))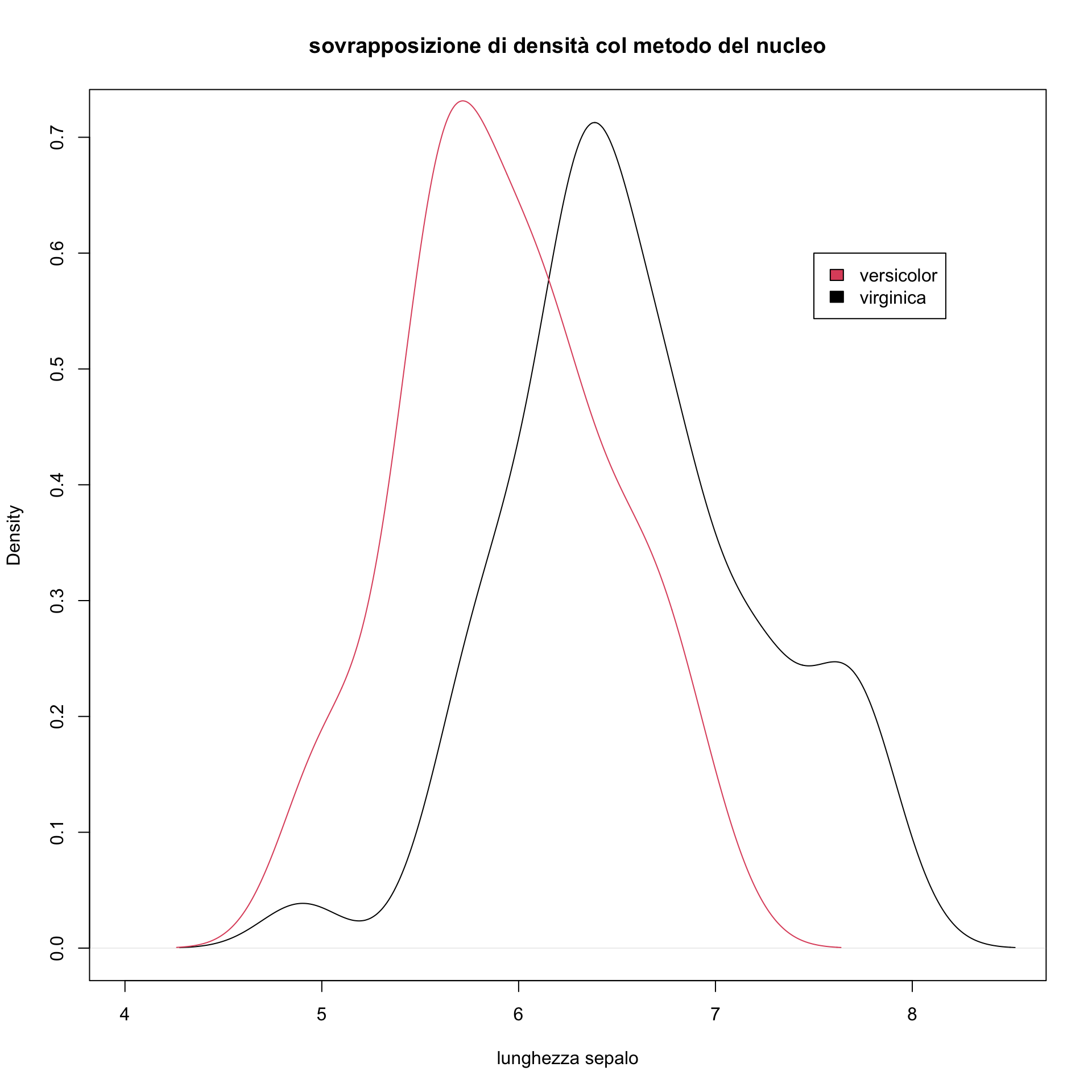
Come si vede, si tratta di un grafico più efficace dell’istogramma per fare confronti. Si ricordi che tali rappresentazioni potrebbero rivelarsi più affidabili con gruppi che contengono un numero di dati sufficientemente elevato (diciamo>100).
Potrebbe esser più opportuno affiancare o sovrapporre le figure. Tuttavia in tal caso gestire grafici multipli con la semplice suddivisione della finestra non è la cosa più opportuna. Vediamo un esempio:
par(mfrow=c(2,2))
hist(iris$Sepal.Length[iris$Species=="versicolor"], freq=FALSE,
xlab="lunghezza sepalo", main="istogramma versicolor")
plot(density(versi), main="densità col metodo del nucleo versicolor")
hist(iris$Sepal.Length[iris$Species=="virginica"], main="istogramma virginica",
border=2, freq=FALSE, density=4.5)
plot(density(virgi), xlim=c(4,8.5), col=2,
xlab="lunghezza sepalo", main="densità col metodo del nucleo virginica")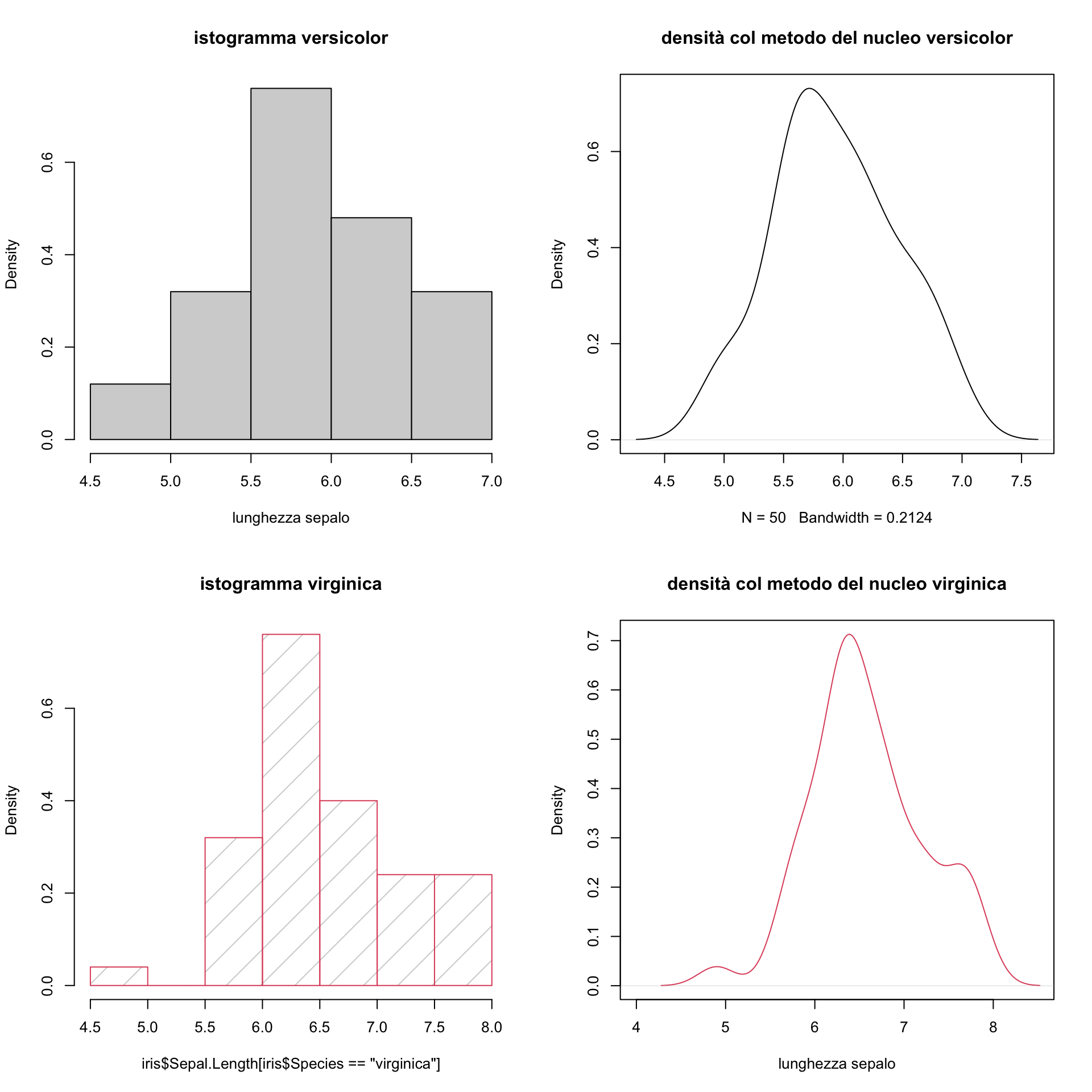
Come si vede il confronto è difficile perchè occorrerebbe adeguare le scale sull’asse orizzontale.
Per fare grafici a più pannelli esistono pacchetti R appositi. Più aventi sarà introdotto, con qualche dettaglio, il più famoso e ampio pacchetto, ggplot2, che fornisce strumenti per ottenere visualizzazioni dei dati molto efficaci.
Ora si introduce, un altro il pacchetto, lattice che contiene varie funzioni che servono a realizzare facilmente grafici multipli (oggetti di tipo trellis). Di seguito un l’esempio con l’istogramma e la densità empirica.
# ci si assicuri di avere scaricato il package lattice
library(lattice)
histogram(~Sepal.Length | Species, data=iris, type="density", layout=c(1,3))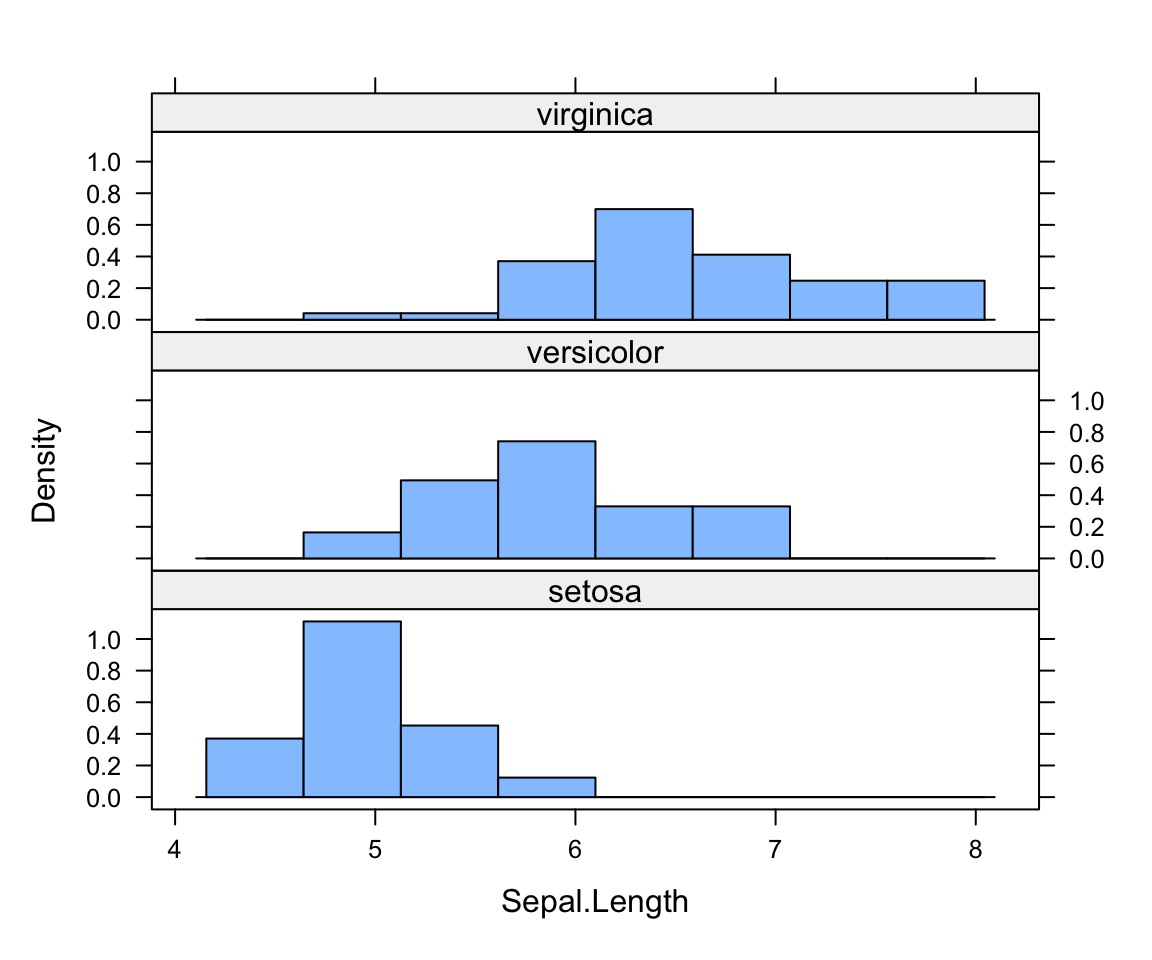
il package lattice contiene molti altri tipi di grafici che possono essere poi organizzati e gestiti in modo opportuno. Si rinvia all’help del pacchetto per dettagli.
5.1.2.4 Il Grafico quantile-quantile
Il confronto dei quantili empirici per corrispondenti valori di \(p\) è uno strumento utile a confrontare due distribuzioni. Se le due distribuzioni sono simili allora i corrispondenti quantili di ordine \(p\) dovrebbero essere gli stessi.
Si può quindi usare il medesimo grafico visto per confrontare quantili empirici e teorici, con la differenza che in questo caso avremo quantili empirici ottenuti per due diversi insiemi di dati.
Si noti che nel caso i due insiemi di dati che si confrontano sono della stessa numerosità \(n\) si tratta semplicemete di un grafico in cui si rappresentano i punti di coordinate corrispondenti ai due vettori ordinati.
Per cui il dato più piccolo in entrambi gli insiemi rappresenta un quantile di pari ordine cioè pari a \(1/n\), il secondo valore ordinato saraà il quantile di ordine \(2/n\) per i due gruppi, e così via.
# Si considerino i dati sulla lunghezza dei sepali per i tipo virginica e versicolor
length(virgi)
length(versi)
# hanno la stessa lunghezza. Il dato più piccolo in entrambi gli insiemi
# rappresenta un quantile di pari ordine cioè pari a 1/50, il secondo valore
# ordinato 2/50 etc.
plot(sort(virgi),sort(versi), pch=19,
main="grafico quantile-quantile")
abline(0,1)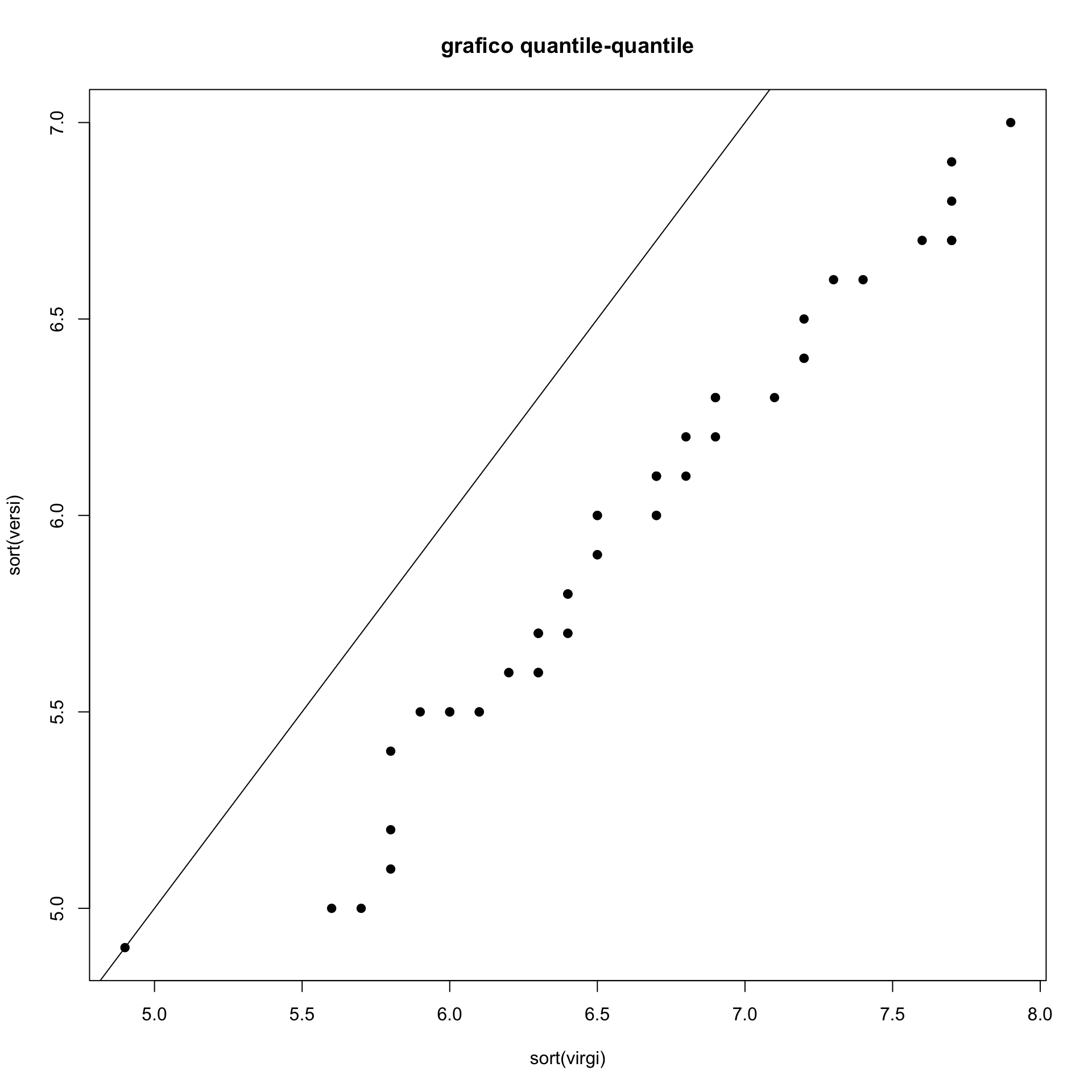
## [1] 50
## [1] 50Se le due distribuzioni fossero uguali dovrebbere esssere disposti lungo la bisettrice. Si vede che non lo sono e in effetti i sepali della specie virginica sono più lunghi.
Tuttavia i dati sembrano disposti lungo una retta che è quasi parallela alla bisettrice. Questo indica che i dati sui sepali della specie virginica pur essendo spostati a destra hanno una distribuzione simile.
Si potrebbe considerare invece della bisettrice una linea che passi fra i rispettivi quartili.
qvirgi<-quantile(virgi,probs=c(.25,.75))
qversi<-quantile(versi,probs=c(.25,.75))
s<-(qversi[2]-qversi[1])/(qvirgi[2]-qvirgi[1])
int<-qversi[1]-s*qvirgi[1]
plot(sort(virgi),sort(versi), pch=19,
main="grafico quantile-quantile")
abline(int,s)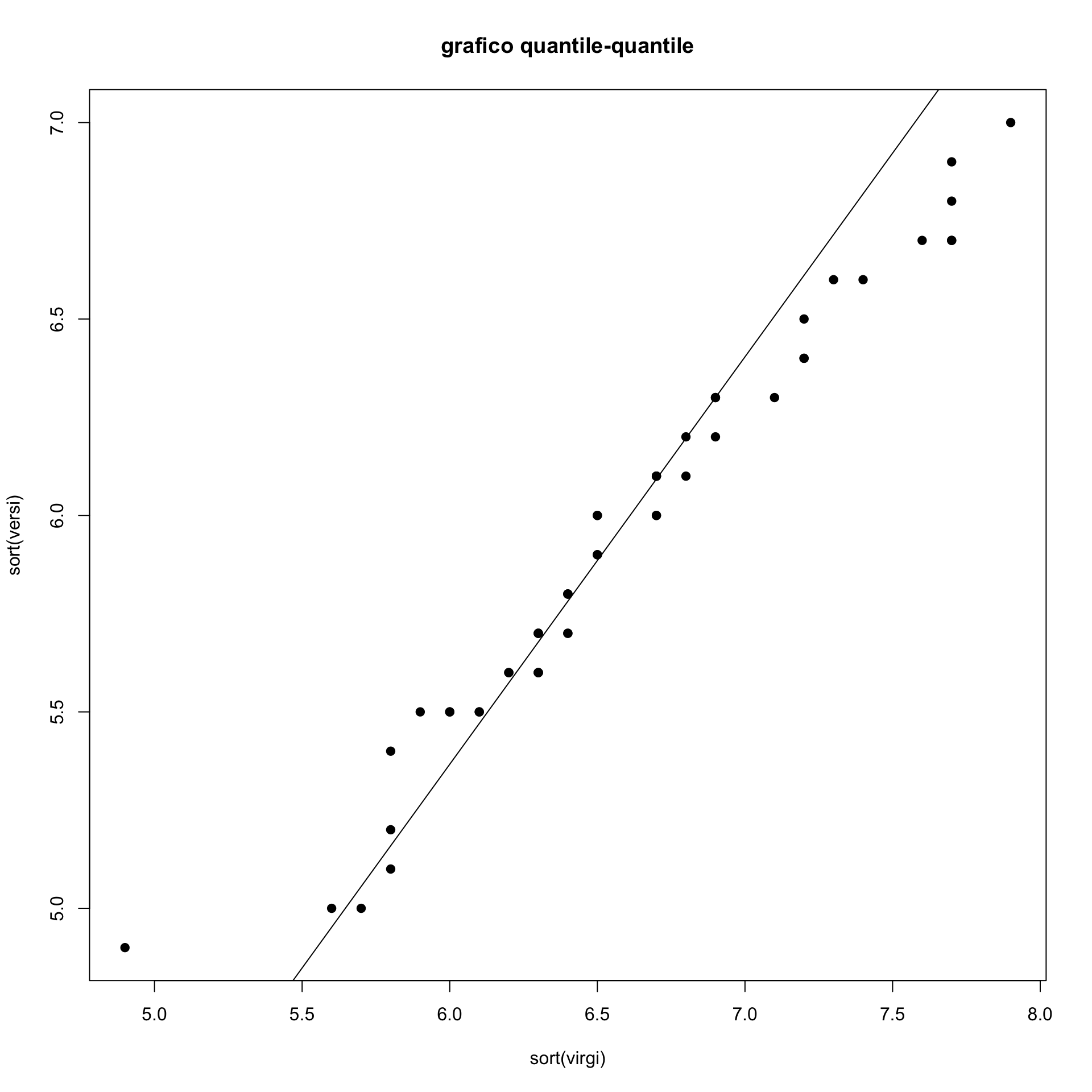
Tale linea di riferimento permette di cogliere lo scostamento che esiste fra i due gruppi.
Ovviamente quello che si è fatto è quanto si otterrebbe con l’uso della funzione qqplot().
 In sostanza:
In sostanza:
- se i punti del qqplot si dispongono su una linea retta le due distribuzioni sono simili come forma.
- se tale retta è parallela alla bisettrice allora le due distribuzioni differiscono in media (ma hanno la stessa forma);
- se tale retta non è parallela allora hanno stessa forma ma diverse media e varianza;
- se i punti non si dispongono lungo una retta allora i due insiemi differiscono anche come forma.
setosa<-iris$Sepal.Length[iris$Species=="setosa"]
qqplot(virgi, setosa )
abline(0,1)
qsetosa<-quantile(setosa,probs=c(.25,.75))
s<-(qsetosa[2]-qsetosa[1])/(qvirgi[2]-qvirgi[1])
int<-qsetosa[1]-s*qvirgi[1]
abline(int,s)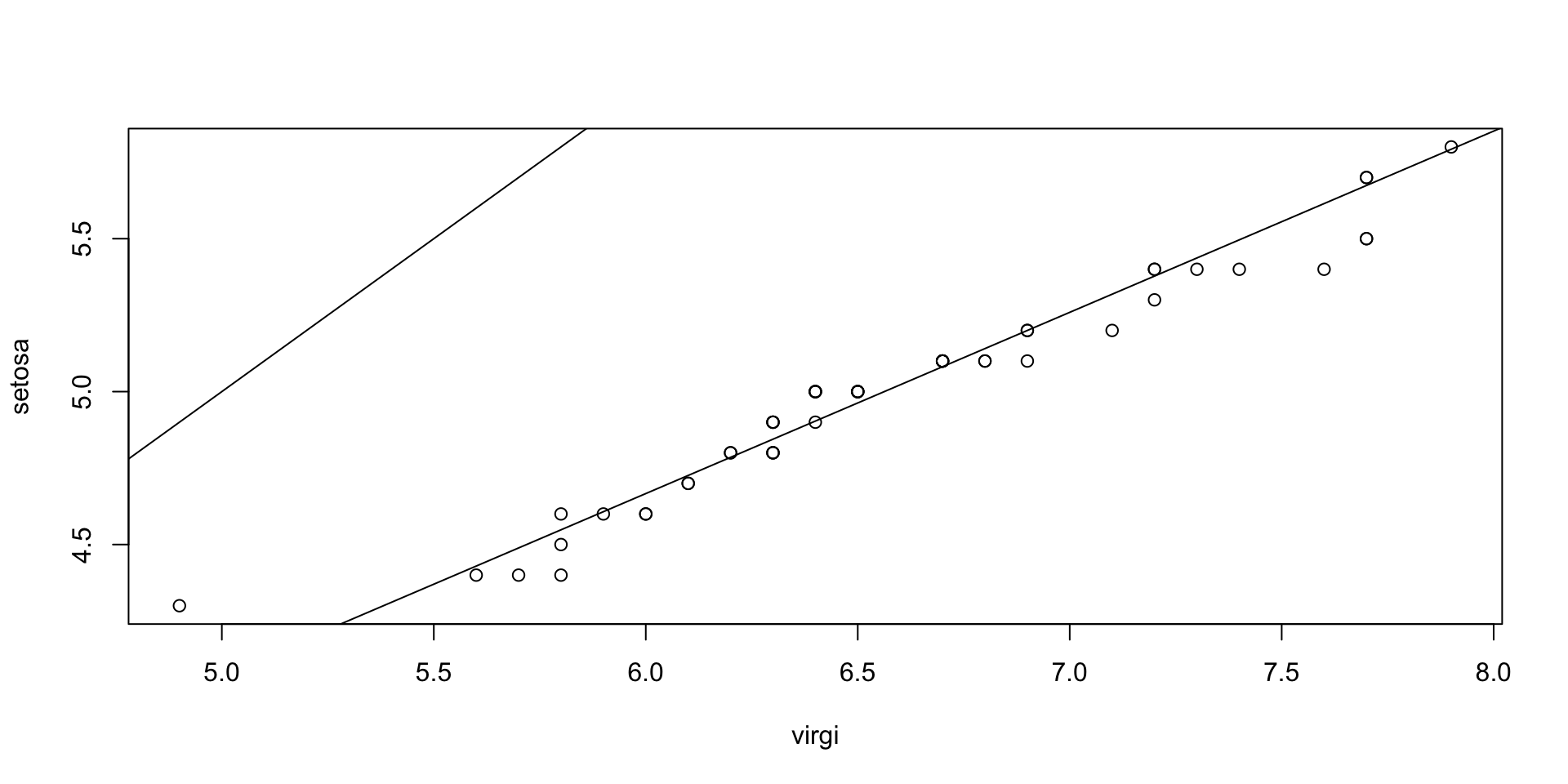
# in questo caso i punti sono lungo una linea ma non parallele alla
# bisettrice. Le distribuzioni sono simili ma con diversa media e varianzaOvviamente il qq-plot può essere ottenuto anche in presenza di gruppi di numerosità diversa. In questo caso vengono calcolati i quantili corrispondenti a una sequenza di valori di \(p\) (che potrebbe corrispondere a quelli dell’insieme meno numeroso)
5.2 Analisi statistica bivariata
L’analisi congiunta di una coppia di variabili, due colonne di un data frame, permette di arricchire enormemente l’analisi dei dati. Agli aspetti già sottolineati con l’analisi di una singola variabile (ovvero sintetizzare sia numericamente che graficamente l’informazione raccolta su un insieme di unità statistiche), se ne aggiungono di nuovi e di maggiore interesse.
In particolare, quello che si vorrebbe valutare è la presenza di relazioni fra le due variabili. Le due variabili possono infatti essere associate nel senso che alcune specifiche coppie di valori (o di modalità) delle due variabili tendono a presentarsi con maggiore frequenza e regolarità. Ad esempio, a valori elevati di una prima variabile quantitativa potrei trovare spesso associati valori elevati di un’altra variabile quantitativa. Oppure, potrei osservare, per i dati di cui si dispone, che la modalità di una variabile qualitativa è spesso associata a una specifica modalità di una seconda variabile qualitativa. Questi aspetti possono emergere solo dall’analisi congiunta di due variabili. Quando questo accade si dirà che le due variabili presentano una qualche forma di associazione.
La presenza di una relazione (associazione) fra le due variabili è potenzialmente di grande interesse perchè la conoscenza di una delle due variabili consente di descrivere o sientetizzare più accuratamente la seconda variabile fino al punto di poter prevedere con minore incertezza i valori (o le modalità) che essa può assumere. L’assenza di relazione (di associazione) fra le variabili, viceversa, è parallelo al concetto di indipendenza, già incontrato nel calcolo delle probabilità, e implica che l’analisi congiunta di due variabili non aggiunge nulla di nuovo e interessante a quello che si era imparato dall’analisi delle variabili prese singolarmente.
In realtà il tema della analisi congiunta di due variabili è stato già introdotto nelle sezioni precedenti quando si è mostrato come si possa ottenere una tabella a doppia entrata analizzando congiuntamente due variabili categoriali (fattori, quindi, o trasformate in fattori attraverso una aggregazione in classi). Ed è stato di fatto introdotto anche quando si è parlato del confronto della stessa variabile quantitativa in più gruppi (ad esempio, i boxplot multipli).
Al fine di considerare gli strumenti per l’analisi di due variabili converrà suddividere il tema a seconda della natura delle variabili. Seppure alcuni concetti generali sono comuni, gli strumenti di analisi e di rappresentazione grafica potranno essere diversi e, come nel caso di una singola variabile, più complessi quando sono coinvolte variabili quantitative.
5.2.1 Due variabili categoriali
Come già illustrato nella prima parte, la sintesi più elementare dei dati per una coppia di variabili è la tabella a doppia entrata.
Riprendiamo l’esempio già visto per i dati AutoBi.
library(insuranceData)
data("AutoBi")
AutoBi$MARITAL<- factor(AutoBi$MARITAL)
levels(AutoBi$MARITAL)<-c("married", "single", "previouslymarried", "previouslymarried")
AutoBi$LOSSclass<-cut(AutoBi$LOSS,breaks=c(0,0.5,2,4,8,1100))
AutoBi$ATTORNEY<- factor(AutoBi$ATTORNEY)
levels(AutoBi$ATTORNEY) = c("yes", "no")
AutoBi$CLMSEX<-factor(AutoBi$CLMSEX)
levels(AutoBi$CLMSEX) <- c("M", "F")
tab1 <- table(AutoBi$ATTORNEY,AutoBi$CLMSEX)
tab1##
## M F
## yes 325 352
## no 261 390##
## M F
## yes 0.4800591 0.5199409
## no 0.4009217 0.5990783##
## M F
## yes 0.5546075 0.4743935
## no 0.4453925 0.5256065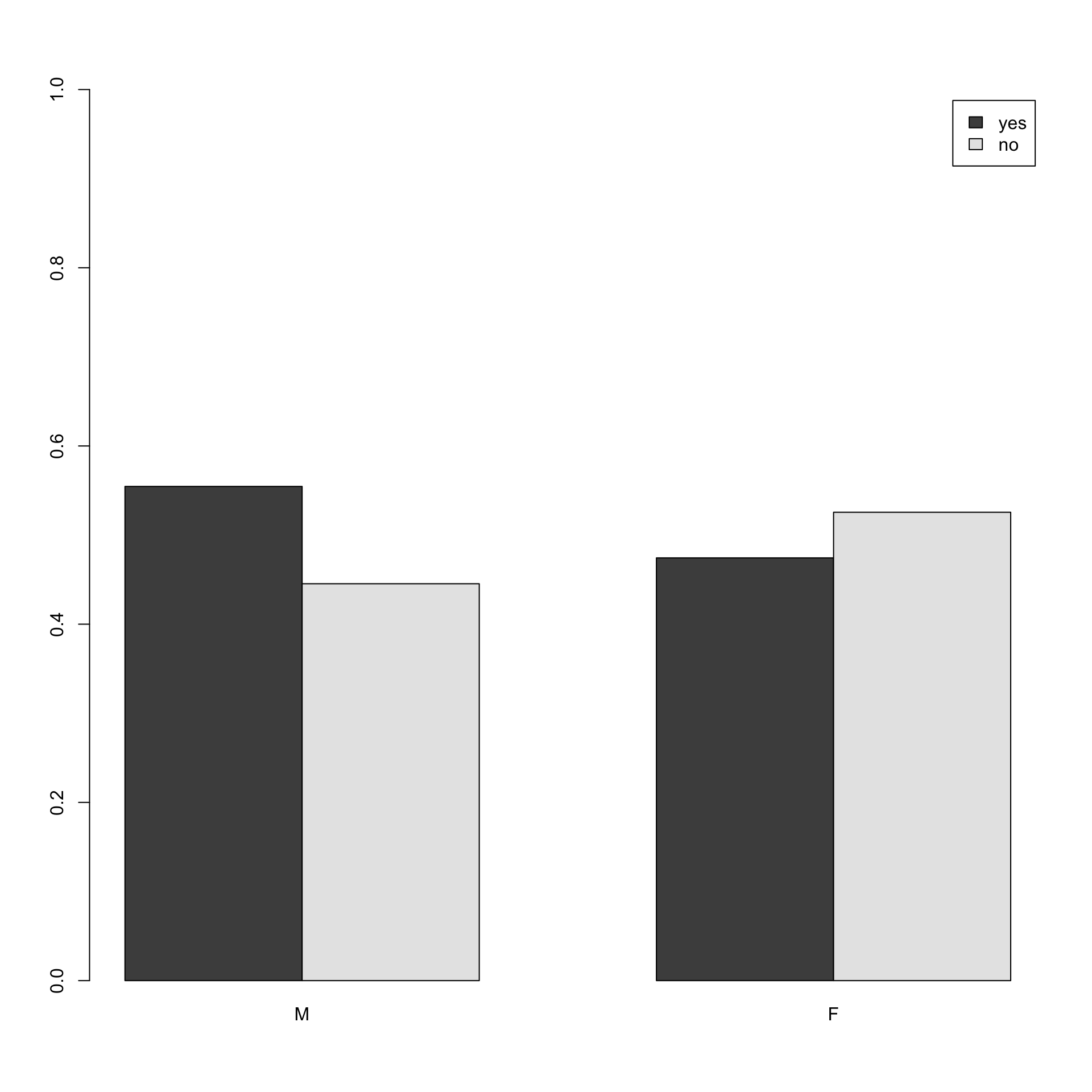
# In questo caso vengono affiancate le due distribuzioni del ricorso
# all'avvocato condizionatamente al sesso
barplot(prop.table(table(AutoBi$ATTORNEY,AutoBi$LOSSclass),2), beside=T,
legend=T, ylim=c(0,1.2))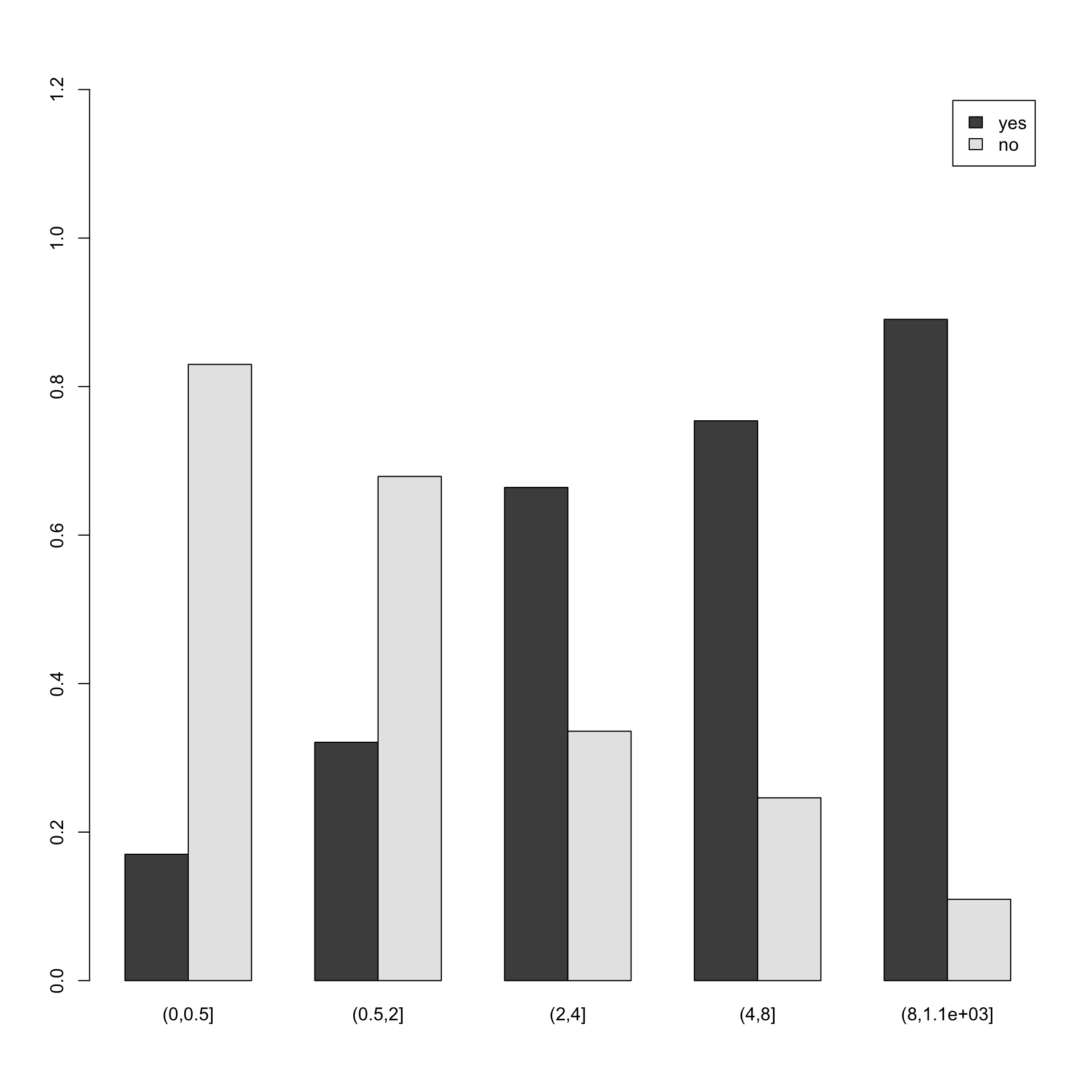
# In questo caso vengono affiancate le due distribuzioni del ricorso
# all'avvocato condizionatamente al danno (trattato in classi come fattore)Come si è già detto, la lettura di una tabella a doppia entrata va spesso fatta avendo in mente il ruolo delle due variabili. In effetti, è utile assegnare, nell’analisi che si svolge, a una delle due variabili il ruolo di variabile risposta (o dipendente): si tratta della variabile di principale interesse. L’altra variabile ha il ruolo di fattore esplicativo ed è detto variabile indipendente o covariata. Di solito questo ruolo è chiaro dal contesto dello studio. In questo caso è utile, ai fini interpretativi, rappresentare la distribuzione della variabile risposta condizionatamente alla variabile esplicativa.
In questo esempio, potrebbe essere interessante capire se vi sono delle tipicità nel ricorso all’avvocato: accade più per gli uomini o è più tipico delle donne? Quindi la variabile ATTORNEY è quella dipendente e verifichiamo se essa cambia condizionatamente al valore assunto dall’altra variabile. Si tratta quindi di confrontare semplici distribuzioni di una variabile dicotomica (Attorney=si, Attorney=no) per i diversi sottoinsiemi identificati dalla modalità di un’altra variabile. Questo è quello che si è fatto con i grafici precedenti dove si confrontavano le distribuzioni del ricorso all’avvocato condizionate all’essere maschio o femmina oppure ad avere avuto una determinata classe di danno.
Se le distribuzioni condizionate sono diverse allora conoscere la variabile indipendente ci consente di prevedere più accuratamente la variabile risposta.
5.2.1.1 La statistica \(X^2\)
Il principio fondamentale da tenere a mente è:
Se le distribuzioni condizionate di una variabile rispetto all’altra sono uguali allora le due variabili sono indipendenti.
Se vi è indipendenza far due variabili non è utile condurre un’analisi congiunta delle stesse poichè tutta l’informazione è contenuta nella distribuzioni marginali.
Il concetto è evidentemente mutuato dal concetto di indipendenza del calcolo delle probabilità e in particolare dalla definizione di indipendenza tra due variabili aleatorie.
Ovviamente, nella pratica sarà piuttosto difficile che si presenti una situazione di perfetta uguaglianza delle distribuzioni condizionate. Per cui risulta utile ottenere delle statistiche che misurino la distanza fra la situazione ideale di indipendenza e quella osservata.
La più nota misura è la statistica \(X^2\). Essa si propone di valutare la distanza fra la tabella a doppia entrata effettivamente osservata e con quello che ci si sarebbe atteso di osservare se vi fosse indipendenza.
Una tabella a doppia entrata per una coppia \((X,Y)\) di variabili le cui modalità sono indicate per riga (la \(X\)) e per colonna (la \(Y\)), ha la seguente forma generale
Le celle, definite dall’incrocio delle righe con le colonne, contengono il numero di casi (\(n_{hk}\)) che presentano le corrispondenti modalità delle due variabili. I valori \(n_{hk}\) sono le frequenze congiunte delle coppie \((x_h,y_k)\) al variare di \(h\)(\(=1,\ldots, H\)) e \(k\)(\(=1,\ldots,K\)).
Le somme per riga, \(n_{h.}={\scriptstyle \sum_{k=1}^{K}}n_{hk}\), e per colonna, \(n_{.k}={\scriptstyle \sum_{h=1}^{H}}n_{hk}\), sono dette anche frequenze marginali di \(x_h\) (di riga) per \(h=1,\ldots,H\) e, rispettivamente, \(y_k\) (di colonna) per \(k=1,\ldots,K\); infine, la numerosità totale è denotata mediante \(n_{}={\scriptstyle\underset{h,k}{\sum}}\,n_{hk}\).
Se vi fosse indipendenza le distribuzioni marginali (di riga) dovrebbero essere tutte uguali alle distribuzioni condizionate della variabile di riga (ovviamente lo stesso varrebbe per la marginale di colonna). Per cui se vi fosse indipendenza, data una generica cella \(h,k\), deve essere \(\forall h,k\) \[\frac{n_{hk}}{n_{h.}}=\frac{n_{.k}}{n}\] quindi la frequenza \(\hat{n}_{hk}\) in caso di indipendenza sarebbe pari a \[\hat{n}_{hk}=\frac{n_{h.}n_{.k}}{n}\].
La statistica \(X^2\) si basa sul confronto fra questi valori e quelli effettivamente osservati. Ovviamente più piccola è tale statistica maggiore è l’indizio che vi sia indipendenza e che le discrepanze osservate debbano attribuirsi solo a scostamenti dovuti al caso.
\[X^2=\sum_{\forall\mbox{cella}}\frac{(n_{hk}-\hat{n}_{hk})^2}{\hat{n}_{hk}}\]
Una valutazione di quanto tale discrepanza sia da ritenersi poco rilevante, e compatibile con fluttuazioni casuali, può essere condotta nell’ambito della statistica inferenziale dove si formula e si valuta rigorosamente una ipotesi di indipendenza fra le due variabili.
In attesa di affrontare tale argomento in modo formale in corsi successivi, si introduce qui, in modo informale, un criterio che consente di giudicare quanto le differenze fra quanto si osserva e quanto ci si aspetta di osservare in caso di indipenedenza siano da ritenersi solo effetto di fluttuazioni casuali:
se effettivamente le due variabili fossero indipendenti, allora il valore della statistica \(X^2\) dovrebbe esser assimilabile a un valore tratto da un particolare tipo di variabile aleatoria nota come variabile \(\chi^2_g\). Essa ha una distribuzione che dipende dal parametro \(g\) che in questo caso è \(g=(H-1)(K-1)\).
Con R si può valutare quanto il valore osservato nella tabella, \(X^2_{oss}\), sia una valore che è plausibile provenga da qualla distribuzione di probabilità. A tal fine si calcola la probabilità di ottenere valori più grandi di \(X^2_{oss}\). Questa probabilità, che è detta \(p\)-value, può essere usato per valutare quanto i dati sono conformi all’ipotesi di indipendenza: se essa è molto piccola (diciamo molto sotto a 0.01) allora non vi sono elementi nei dati a sostegno della indipendenza fra le due variabili.
Ad esempio:
# riconsideriamo la tabella per sesso e ricorso
tab1 # all'avvocato
tot<-marginSums(tab1)
mr<- margin.table(tab1,1) # Marginale di riga (variabile avvocato)
mc<- margin.table(tab1,2) # Marginale di colonna (variabile sesso)
hatn<- mr%*%t(mc)/tot # tabella delle frequenze teoriche di indipenedenza
hatn
X2<-sum((tab1-hatn)^2/hatn) # calcolo X^2
X2
pchisq(X2,1, lower.tail = FALSE) # calcolo la probabilità di ottenere un valore maggiore di
# quello ottenuto per X^2 in un chi-quadrato con 1 gdl##
## M F
## yes 325 352
## no 261 390
##
## M F
## yes 298.7364 378.2636
## no 287.2636 363.7364
## [1] 8.430048
## [1] 0.003690706# Esiste la funzione R che svolge questi calcoli
chisq.test(tab1, correct=FALSE)
# Come si vede fornisce gli stessi risultati. Il parametro correct=FALSE
# evita che venga applicata una correzione al calcolo della differenze fra valori
# osservati e valori di indipendenza nota come correzione per continuità##
## Pearson's Chi-squared test
##
## data: tab1
## X-squared = 8.43, df = 1, p-value = 0.003691# si provi anche con la tabella ricorso all'avvocata e danno accertato (in classi)
tab2<-table(AutoBi$ATTORNEY,AutoBi$LOSSclass)
tab2
chisq.test(tab2)##
## (0,0.5] (0.5,2] (2,4] (4,8] (8,1.1e+03]
## yes 49 104 263 147 122
## no 239 220 133 48 15
##
## Pearson's Chi-squared test
##
## data: tab2
## X-squared = 342.89, df = 4, p-value < 2.2e-16La probabilità, che denominata \(p\)-value, nei due esempi mostrati è abbastanza piccola, e in particolare nel caso della relazione fra ricorso all’avvocato e danno esso è così piccola da poter sostenere che se vi fosse indipendenza è del tutto implausibile osservare quei dati e pertanto essi non danno alcun sostegno a tale ipotesi.
Anche nel caso del sesso e ricorso all’avvocato, il \(p-value\) è sufficientemente piccolo da poter concludere che i dati non danno molto sostegno all’ipotesi di indipendenza. Tuttavia, non è estremamente piccolo come nel caso precedente: la associazione fra le due variabile è presente ma è meno marcata che nel caso precedente. Questo è in accordo con quanto si osserva nei grafici.
5.2.1.2 Le tabelle \(2\times2\) e l’odds ratio
Gli odds-ratio forniscono una misura semplice e immediatamente comprensibile riguardo al presenza di associazione in una tabella di frequenze congiunte quando le due variabili sono dicotomiche (tabelle \(2\times2\)).
Consideriamo due variabili dicotomiche: \(Y=\{occupato,disoccupato\}\) e \(X=\{laureato, non laureato\}\) osservate per un gruppo di 1000 persone. La tabella delle frequenze congiunte sia
| Y | occupato | non occupato | |
|---|---|---|---|
| X | |||
| laureato | 210 | 105 | |
| non laureato | 353 | 332 |
In termini di odds-ratio (rapporto fra gli odds) la tabella si può sintetizzare con il rapporto tra laureati e non laureati tra gli occupati \(210/353=0.59\) e laureati e non laureati tra i non occupati \(105/332=0.31\), il cui risultato è \(1.88\). Gli odds sono espressioni simili ai rapporti di scommessa (del tipo il cavallo fulmine è dato 1 a 3) per cui si potrebbe dire, in questo caso, che il rapporto fra laureati e non laureati (tra i non occupati) e circa di 1 a 3 (1 laureato ogni 3 non laureati).
Si noti che il valore dell’odds-ratio si ottiene anche facendo il rapporto fra il prodotto dei valori che nella tabella sono sulla diagonale principale e il prodotto dei valori sulla diagonale secondaria \((210\times332)/(105\times353)\). Esso è infatti anche detto rapporto dei prodotti incrociati.
Un rapporto vicino ad 1 indica una scarsa associazione tra le variabili, (infatti se il rapporto è esattamente 1 ciò implica che le distribuzioni condizionate sono uguali e vi è quindi indipendenza fra i caratteri in gioco). Quanto più il valore è distante da 1 tanto maggiore è l’associazione fra le due variabili. Un risultato maggiore di 1 come nel nostro caso indica una associazione positiva tra la modalità occupato e quella laureato (ovvero la frequenza osservata per questa coppia di modalità è maggiore della frequenza che si osserverebbe se vi fosse indipendenza).
Chiaramente, il reciproco di 1.88 (pari circa a 0.53) misura un’associazione che è esattamente della stessa forza (tale valore si otterrebbe difatti se si permutassero le righe della medesima tabella). L’allontanamento da 1 (cioè dall’indipendenza) dell’odds ratio va letto quindi in maniera diversa se il valore è maggiore o minore di 1 poiché evidentemento esso non può assumere valori negativi. In sostanza in una tabella ove risulti un odds ratio pari a 5 (che indica una forte associazione) è presente un’associazione del tutto equivalente ad una tabella per cui risulti un odds ratio pari a 0.2. Si noti infine che è possibile definire correttamente il rapporto solo se tutte le caselle della tabella hanno frequenze superiori a 0.
Per rendere più leggibili queste quantità spesso conviene passare ai logaritmi: il log-odd-ratio è quindi 0 se le variabili sono indipendenti, ed assume comunque lo stesso valore assoluto comunque si permutino righe e colonne della stessa tabella.
Consideriamo ancora i dati già analizzati su Ricorso all’avvocato e Genere, e calcoliamo odds-ratio e log-odds-ratio.
##
## M F
## yes 325 352
## no 261 390# calcoliamo direttamente il rapporto dei prodotti incrociati
OR<-(tab1[1,1]*tab1[2,2])/(tab1[1,2]*tab1[2,1])
OR## [1] 1.379637## [1] 0.3218203Si noti come il valore di OR e di LOR sono abbastanza diversi, rispettivamente, da 1 e da 0, e forsniscono indicazione della assenza di indipendenza.
5.2.2 Una variabile quantitativa condizionata a un fattore
Come si è già detto, è spesso opportuno valutare il ruolo delle variabili coinvolte e chiedersi se, nello studio congiunto, si ritiene che si possa distinguere una delle due variabili come variabile risposta da analizzare condizionatamente a una seconda. Nel caso in questione si supporrà che la variabile risposta sia quella quantitativa. La seconda variabile, categoriale, è quindi la variabile di condizionamento ed essendo un fattore qualitativo esso identifica dei gruppi in corrispondenza delle diverse modalità del fattore. In questo caso si tratta quindi di studiare le distribuzioni della variabile risposta in ciascuno di questi gruppi e verificare se differiscano e quanto e come differiscano.
Resta fermo quanto già osservato, ovvero:
se le distribuzioni della variabile quantitativa per ciascun gruppo definito dal fattore (le distribuzioni condizionate a ciascuna modalità del fattore) sono simili allora l’analisi bivariata non fornisce elementi di interesse.
Se le due variabili coinvolte fossero indipendenti allora ci aspetteremmo che le distribuzioni condizionate siano uguali.
Si denoti con \(Y\) la variabile risposta quantitativa e con \(X\) il fattore qualitativo, con \(G\) modalità, che definisce i gruppi di valori: si vogliono analizzare la variabili \(Y|X=x_g\) con \(g=1,2,\dots,G\).
Poichè \(Y\) è quantitativa noi abbiamo molti modi per studiare somiglianze e differenze fra le distribuzioni condizionate.
- Si potrebbe ritenere che le distribuzioni sono tutte uguali tra loro come forma e che si differenzino solo per quanto riguarda la tendenza centrale (misurata, ad esempio, dalla media o dalla mediana).
- Le distribuzioni potrebbero avere la stessa forma (ad esempio, simmetrica e con curtosi simile a quella della gaussiana) ma sono diverse sia la tendenza centrale che la variabilità.
- Le due distribuzioni potrebbero essere diverse tra loro in modo più radicale (in un gruppo abbiamo distribuzioni simmetriche in un altro sono asimmetriche).
Alcune delle rappresentazioni grafiche già introdotte sono idonee a valutare in una analisi esplorativa quale delle situazioni sopra elencate possa essere più plausibile. Nei diversi casi si può ricorrere a strumenti diversi per la sintesi dei dati.
Se in particolare il fattore ha solo due modalità (e definisce quindi solo due gruppi di dati per la variabile \(Y\)) strumenti grafici come il confronto della funzione di ripartizione empirica dei due gruppi o il grafico quantile-quantile possano rivelarsi utili.
Se \(X\) ha più di due modalità e vi sono più gruppi risulta utile l’analisi grafica con box-plot multiplo o il confronto degli istogrammi (o delle funzioni di densità empirica).
Resta da aggiungere, infine, la possibilità di ricorrere a strumenti analitici per rappresentare le diverse situazioni o a strumenti tipici della inferenza statistica (in particolare, i cosiddetti test statistici) per verificare se le differenze riscontrate nelle distribuzioni condizionate siano da ritenersi solo dovute a oscillazioni causali e siano quindi compatibili con l’ipotesi che tali distribuzioni siano uguali tra loro. In questo ultimo caso il ricorso a analisi condizionata non aggiunge informazioni ed sufficinete guardare alla variabile marginale \(Y\).
Ciascuno degli strumenti statistici ha delle funzioni in R che consentono facilmente di ottenere le informazioni rilevanti.
Prima di presentarne alcuni conviene introdurre alacune proprietà elementari che riguardano l’analisi di una variabile quantitativa osservata in più gruppi.
5.2.3 La scomposizione della devianza
Un’importante proprietà della media aritmetica è l’associatività: .
Si consideri una matrice di dati, esemplificata nello schema seguente, in cui \(n\) osservazioni su una variabile quantitativa \(Y\) sono suddivise, secondo le modalità di una variabile categoriale \(X\), in \(G\) gruppi di numerosità \(n_1,\ldots,n_G\) in relazione alle modalità di \(X\). \[\begin{equation*} \begin{matrix} & x_1 & x_2 & \cdots & x_g & \cdots & x_G \\\hline 1 &y_1^{(1)} & y_1^{(2)} & \cdots & y_1^{(g)} & \cdots & y_1^{(G)} \\ 2 &y_2^{(1)} & y_2^{(2)} & \cdots & y_2^{(g)} & \cdots & y_2^{(G)} \\ \vdots &\vdots & \vdots & & \vdots & & \vdots \\ i &y_i^{(1)} & y_i^{(2)} & \cdots & y_i^{(g)} & \cdots & y_i^{(G)} \\ \vdots &\vdots & \vdots & & \vdots & & \vdots \\ n_1 &y_{n_1}^{(1)} & \vdots & & \vdots & & \vdots \\ \vdots & & \vdots & & \vdots & & \vdots \\ n_G & & \vdots & & \vdots & & y_{n_G}^{(G)} \\ \vdots & & \vdots & & \vdots & & \\ n_2 & & y_{n_2}^{(2)} & & \vdots & & \\ \vdots & & & & \vdots & & \\ n_g & & & & y_{n_g}^{(g)} & & \\ \end{matrix} \end{equation*}\]
Si indichi con \(m\) la media complessiva degli \(n=\sum_{g=1}^G n_g\) valori, cioè la media di tutti i dati in cui ignoro la suddivisione in gruppi, \[ m=\frac{1}{\sum_{g=1}^G n_g}\sum_{g=1}^G\sum_{i=1}^{n_g} y_i^{(g)}, \] sia poi \(m_g=\frac{1}{n_g}\sum_{i=1}^{n_g}y_i^{(g)}\) la media specifica per il gruppo \(g\) (è la media condizionata \(M(Y|X=x_g)\) ), si ha allora \[\begin{equation} \label{eq:medass} m=\sum_{g=1}^G \frac{n_g}{n} m_g, \end{equation}\] infatti \[\begin{equation*} m = \frac{1}{n}\sum_{g=1}^G \sum_{i=1}^{n_g} y_i^{(g)} = \sum_{g=1}^G \frac{n_g}{n} \frac{\sum_{i=1}^{n_g} x_i^{(g)}}{n_g} = \sum_{g=1}^G \frac{n_g}{n} m_g. \end{equation*}\] Vale la pena di osservare che questa proprietà si estende anche alle medie che possono essere espresse come medie aritmetiche di opportune trasformazioni dei dati. Sono esempi la media quadratica, che è media aritmetica dei dati trasformati secondo \(f(y)=y^2\) e, in generale, le medie di ordine \(s\).
Lievemente più complicato è esprimere la devianza in funzione della devianza all’interno dei gruppi.
Partendo dalla definizione di devianza \(DEV_{TOT}\) della totalità dei dati, che possiamo scrivere \[\begin{eqnarray*} DEV_{TOT} & = & \sum_{g=1}^G \sum_{i=1}^{n_g} (y_i^{(g)}-m)^2 \\ & =& \sum_{g=1}^G \sum_{i=1}^{n_g} (y_i^{(g)}-m_g+m_g-m)^2. \end{eqnarray*}\] Sviluppando il quadrato si ha \[\begin{eqnarray*} DEV_{TOT} = \left[\sum_{g=1}^G \sum_{i=1}^{n_g} (y_i^{(g)}-m_g)^2\right. &+& \sum_{g=1}^G \sum_{i=1}^{n_g}(m_g-m)^2 + \\ &+& \left. 2 \sum_{g=1}^G (m_g-m) \underbrace{\sum_{i=1}^{n_g} (y_i^{(g)}-m_g)}_{=0\;\forall g} \right] \end{eqnarray*}\] (dove \(\sum_{i=1}^{n_g} (y_i^{(g)}-m_g)=0\) per una proprietà della media aritmetica) e quindi \[\begin{equation} DEV_{TOT}= \underbrace{\sum_{g=1}^G \sum_{i=1}^{n_g}(y_i^{(g)}-m_g)^2}_{DEV_{\text{within}}} + \underbrace{\sum_{g=1}^G \frac{n_g}{n} (m_g-m)^2}_{DEV_{\text{between}}}. \label{eq:vargruppi} \end{equation}\] La Devianza totale \(DEV_{TOT}\) (complessiva) è cioè pari alla devianza all’interno dei gruppi (devianza within o entro) a cui va sommata la media pesata dei quadrati degli scostamenti delle medie di gruppo dalla media generale (devianza between o fra).
La prima parte è una misura di quanto siano dispersi i valori all’interno di ciascun gruppo, la seconda di quanto siano dispersi i gruppi tra di loro. Ciò risulta più chiaro se si guarda ai due casi limite: se \(DEV_{TOT}=DEV_{\text{within}}\) allora \(m_g=m\) per ogni \(g=1,\ldots G\), cioè non vi è differenza in media tra i gruppi; mentre \(DEV_{TOT}=DEV_{\text{between}}\) quando \(v_g=0\) per ogni \(g=1,\ldots G\), cioè quando tutte le osservazioni all’interno di uno stesso gruppo coincidono.
Si noti che data la relazione
\[DEV_{TOT}=DEV_{within}+DEV_{between}\] Si può valutare quanto i gruppi spieghino della devianza complessiva guardando al rapporto
\[0 \leq \frac{DEV_{between}}{DEV_{TOT}} = \eta^2 \leq 1\]
L’indice \(\eta^2\) (detto rapporto di correlazione) è quindi un indice che se vicino a 1 indica che i dati nei diversi gruppi definiti dalle modalità del fattore sono poco variabili e che la variabilità è in gran parte legata alle differenze fra le medie dei gruppi.
5.2.4 L’uguaglianza dei valori centrali dei diversi gruppi (ANOVA)
La scomposizione della devianza vista sopra è alla base di un’importante statistica che si usa per verificare, usando i metodi dell’inferenza statistica, l’ipotesi che le medie dei diversi gruppi siano uguali.
In questo caso, per usare i metodi dell’inferenza, è necessario sepcificare un opportuno modello statistico che si suppone abbia generato i dati e l’ipotesi riguarderà i parametri i tale modello.
In particolare si suppone che i dati provengano da gaussiane indipendenti \(\mathbb{N}(\mu_g,\sigma^2)\) ove \(g=1,2,\dots,G\) identifica i gruppi identificati da un fattore categoriale con \(G \geq 3\) modalità. Si dispone quindi di campioni casuali semplici di numerosità \(n_g\) da ogni gruppo con \(n=\sum_{g=1}^k n_g\). Si assume anche che in ciascun gruppo la varianza sia sempre la stessa e pari a \(\sigma^2\) (assunzione di omoschedasticità).
Si tratta di valutare le diverse medie dei gruppi siano sufficientemente diverse da poter escludere che questo accada per effetto del caso.
A tal fine si calcola una statistica costituita dal rapporto tra la \(Dev_{between}\) e la \(DEV_{TOT}\) divise per rispettivamente per \(G-1\) e \(n-G\) ovvero
\[ S=\frac{DEV_{between}/(K-1)}{DEV_{within}/(n-K)}\]
Tale statistica, dovrebbe avere valori bassi se le medie sono uguali tra loro e le differenze sono imputabili al caso. Se questo è vero tale statistica equivale a un numero casuale proveniente da una distribuzione di probabilità nota come \(F\) di Fisher. In particolare è distribuita come una \(F_{G-1,n-G}\) in cui \(G-1\) e \(n-G\) sono i parametri di tale distribuzione (e sono detti gradi di liberta rispettivamente del numeratore e del denominatore).
Se i valori di tale statistica sono molto elevati allora è poco plausibile che i dati provengano da tale distribuzione e questo implica che i dati diano poco supporto alla ipotesi che le medie siano tutte uguali tra loro. A tal fine, si può quindi calcolare la \(Pr(F > S)\), questa probabilità è il cosidetto il \(p\)-value. Quanto più questa probabilità è piccola tanto meno sarà plausibile che i dati osservati siano coerenti con l’ipotesi che tutte le medie sono uguali tra loro (i dati tendono a segnalarci che almeno una media è diversa dalle altre).
In inferenza statistica tale procedura è nota come Analisi della Varianza o ANOVA. Essa è di grande rilievo in numerosissimi contesti applicativi. Esistono versioni di tale metodo che richiedono assunzioni meno restrittive, ma si rinvia per esse, come per i dettagli su l atecnica inferenziale dell’ANOVA, a corsi di statistica successivi, più avanzati su temi di statistica inferenziale.
Può essere però interessante vedere come usare tale procedura con R usando la funzione aov().
#Consideriamo ancora i dati di iris. In essi la variabile Species definiva 3 gruppi
data(iris)
testanova<-aov(Sepal.Length~Species, data=iris) # usiamo la notazione già vista con la variabile
# di interesse come variabile dipendente dal fattore
summary(testanova)## Df Sum Sq Mean Sq F value Pr(>F)
## Species 2 63.21 31.606 119.3 <2e-16 ***
## Residuals 147 38.96 0.265
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1La seconda colonna (Sum Sq) contiene le due devianze rilevanti (la devianza between e quella within)
# Si noti che la loro somma è pari alla devianza della variabile di interesse, infatti
# calcoliamo la varianza totale dell lunghezza del sepalo
n<-length(iris$Sepal.Length)
Devtot<-var(iris$Sepal.Length)*(n-1)
Devtot## [1] 102.16835.3 Analisi di due variabili quantitative
L’analisi congiunta di due variabili quantitative, diciamo \((X,Y)\), si propone ancora di valutare se siano associate ed eventualmente di capire che forma assume tale associazione.
5.3.1 Il diagramma di dispersione
Dal punto di vista della rappresentazione grafica, i dati sulle due variabili si presenteranno come coppie di valori numerici \((x_i,y_i)\) (con \(i=1,2,\dots,n\)). Essi possono essere rappresentati direttamente come punti con coordinate \(x_i,y_i\), possiamo usare quindi in R il comando plot per ottenere un grafico che in questo contesto viene denominato diagramma di dispersione (in inglese si usa il termine scatterplot).
Si considerino i dati nel dataframe whiteside nel package MASS. Contiene tre variabili: Gas, Temp e Insul. Si riferiscono a dati raccolti su abitazioni e Temp è la temperatura esterna (in gradi), Gas (in mc) è il consumo di gas per il riscaldamento dell’abitazione, mentre Insul è un fattore che registra se i dati si riferisco a misurazioni prese prima o dopo un intervento di isolamento termico della struttura.
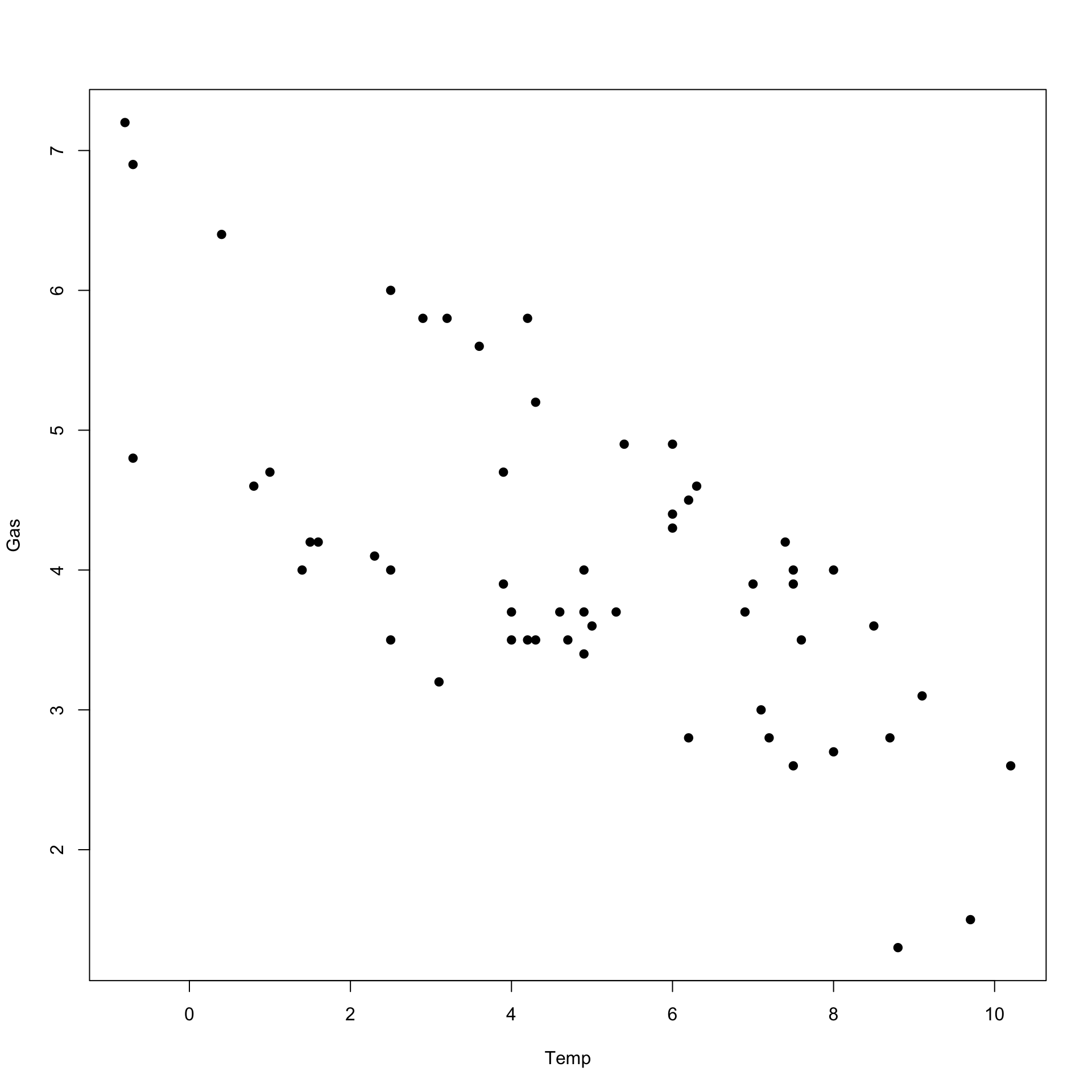
Come era facile attendersi la nuvola di punti è orientata chiaramente e delinea una relazione negativa fra le due variabili.
5.3.2 Le misure di relazione lineare
Per valutare la tendenza delle due variabili a fornire valori che siano associati (positivamente o negativamente) così che, nel caso di relazione positiva, se la prima variabile mostra valori piccoli, ad esempio, anche la seconda tendenzialmente avrà valori piccoli, e lo stesso vale per valori elevati.
È piuttosto evidente nei grafici visti sopra che la nuvola di punti tenda a disporsi lungo una retta così da configurare una tendenziale relazione lineare fra le due variabili. Questa tendenza è misurata dalla covarianza definita come:
\[cov(X,Y)=\sum_{i=1}^n\frac{(x_i-M(x))(y_i-M(y))}{n}\]
Il numeratore dell’espressione è detto codevianza.
Vale per la covarianza lo stesso discorso fatto a suo tempo per la varianza: se essa viene calcolata nell’ambito di un approccio inferenziale, ovvero essa non è semplicemente un indice descrittivo di relazione lineare fra le due variabili, ma assume il ruolo di stimatore della varianza di una popolazione infinita da cui è tratto un campione casuale, si conviene di usare la divisione per \(n-1\). Vedremo che in R la specifica funzione per calcolare la covarianza,
utilizza la divisione per \(n-1\)
Si noti che la covarianza sopra definita, se divisa per \(n\), può essere anche calcolata come: \(cov(X,Y)=M(XY)-M(X)*M(Y)\)
La covarianza assume valori positivi o negativi a seconda che la nuvola di punti tenda a disporsi lungo una retta orientata positivamente o negativamente. Può essere nulla se tale tendenza è assente. Si noti inoltre che \(cov(X,Y)=cov(Y,X)\).
Non è facile valutare il grado della covarianza in quanto il suo valore assoluto dipende dall’unità di misura delle variabili. Per tale motivo è utile ricordare che, in conseguenza della disuguaglianza di Cauchy-Schwartz, si ha
\[cov(X,Y)^2\leq var(X)var(Y)\] che equivale a
\[\begin{equation} \begin{split} -\sqrt{(var(X)var(Y} &\leq cov(X,Y) \leq \sqrt{(var(X)var(Y))} =\\ -sqm(X)sqm(Y) &\leq cov(X,Y) \leq sqm(X)sqm(Y) \end{split} \end{equation}\]
Se quindi si dividono tutti i membri della doppia disuguaglianza sopra per \(sqm(X)sqm(Y)\) si ha
\[-1 \leq \frac{cov(X,Y)}{sqm(X)sqm(Y)}= r(X,Y) \leq 1\] L’espressione \[\frac{cov(X,Y)}{sqm(X)sqm(Y)}\] è quindi la covarianza standardizzata (si noti che si arriva alla stessa espressione se si calcola la covarianza tra le due variabili standardizzate). Tale quantità è nota come coefficiente di correlazione lineare fra le due variabili \(X\) e \(Y\) di solito denotato con \(r(X,Y)\).
Esso è di più agevole interpretazione rispetto alla covarianza: infatti se esso è pari a 1 (o a -1) fra le due variabili vi è una perfetta relazione lineare (la nuvola di punti è perfettamente allineata lungo una retta). In R le funzioni cov()e cor() calcolano ripettivamente le covarianze e il coefficiente di correlazione lineare. La covarianza è calcolata come codevianza diviso per \(n-1\).
# proviamo a calcolare la covarianza e il coeffficiente di correlazione per i dati whiteside
n<-length(Temp)
codev<-sum((Temp-mean(Temp)*(Gas-mean(Gas))))
covar<-codev/n
print(paste("covarianza calcolata dividendo per n", covar))
correl<-codev/(sd(Gas)*sd(Temp))
print(paste("coefficiente di correlazione lineare",correl))## [1] "covarianza calcolata dividendo per n 4.875"
## [1] "coefficiente di correlazione lineare 85.017532033662"# usiamo ora le funzioni di R
cov(Temp,Gas) # si noti che è diverso da quello sopra
# esso è infatti uguale a
codev/(n-1)## [1] -2.194
## [1] 4.9636365.3.3 La regressione lineare semplice
La covarianza e la correlazione misurano la tendenza delle due variabili ad essere associate linearmente. Tuttavia, come già discusso nel caso di due variabili categoriali, risulta di maggiore interesse il caso in cui le due variabili coinvolte hanno nell’analisi un ruolo diverso. Una delle due variabili, la variabile risposta, viene analizzata condizionatamente all’altra (la covariata o variabile indipendente).
L’interesse è quindi nel riuscire a fornire sintesi più efficaci della variabile risposta, diciamo \(Y\), quando si conosca il valore della covariata \(X\). Visto in altri termini, si tratta di capire se conoscere una variabile possa aiutare a prevedere l’altra con minore incertezza.
Per sintetizzare una variabile è spesso utile considerare la sua media e, in tal caso, una misura dell’incertezza è data dalla varianza (si ricordi che la media è il valore che rende minima la somma dei quadrati degli scarti da essa).
Si immagini quindi di voler ottenere per ogni possibile valore di \(X=x\) la media di \(Y\). Essa è la media di \(Y\) condizionata ad \(x\), la si denoti con \(M(Y|x)\).
Quest’ultima è in definitiva una funzione di \(x\) e \(M(Y|x)=f(x)\) è detta funzione di regressione.
Per poter valutare tale funzione si dovrebbe disporre di un insieme di dati su \(Y\) in corrispondenza di ogni possibile valore \(x\). Essendo \(X\) una variabile quantitativa essa assume presumibilmente molti valori distinti e quindi nella realtà di solito non si osservano più valori di \(Y\) per ogni \(x\) quindi non si è in grado di calcolare \(M(Y|x)\) a partire dai dati (a meno che non si trasformi \(X\) in un fattore con un raggruppamento in classi con il consueto problema di perdere informazione).
Una semplice strategia per ottenere la funzione di regressione \(M(Y|x)=f(x)\) è quella di supporre che tale funzione sia molto semplice: ad esempio, si può ipotizzare che le medie condizionate di \(Y\) per ogni \(x\) siano lungo una retta. Si assume quindi che la funzione di regressione sia lineare,cioè: \[M(Y|x)=f(x)=\beta_0+\beta_1x\] Per cui cerchiamo una retta, con intercetta pari a \(\beta_0\) e coefficiente angolare \(\beta_1\), lungo la quale si immagina siano collocate le medie condizionate.
I valori delle due variabili in generale si discosteranno dalla retta, per cui \(\forall i\) si osserverà, con riferimento alla variabile \(Y\), lo scostamento di un valore \(y_i\) da quella che dovrebbe essere la media condizionata \(M(Y|x_i)=\beta_0+\beta_1 x_i\).
Sembra sensato cercare quindi una retta, ovvero due valori \(\beta_0\) e \(\beta_1\), per i quali tali scostamenti siano nel complesso piccoli. Vi sono molti modi di misurare lo scostamento complessivo fra valori osservati e retta. Tuttavia una scelta ragionevole, avendo immaginato che sulla retta ci sono le medie, sarebbe quello di richiedere che per gli scostamenti valga la proprietà già enunciata per una media aritmetica: cerchiamo la retta per cui la somma degli scostamenti al quadrato risulti più piccola possibile, ovvero che sia minima \(\sum_{i=1}^n(y_i-f(x_i))^2\).
5.3.3.1 La retta di regressione dei minimi quadrati
Definiamo con \(\hat{y}_i\) i valori su una retta in corrispondenza dei dati osservati \(x_i\) per la variabile indipendente. I valori \(y_i-\hat{y}_i\) sono detti residui.
Il principio appena enunciato, detto dei minimi quadrati, richiede quindi che definita la funzione \[L(\beta_0,\beta_1)=\sum_{i=1}^n(y_i-f(x_i))^2=\sum_{i=1}^n(y_i-\beta_0-\beta_1 x_i)^2\] che rappresenta la somma dei quadrati dei residui, e che si cerchino i valori \(\beta_0\) e \(\beta_1\) per cui risulti minima \(L(\beta_0,\beta_1)\). Tali valori forniscono i coefficienti della retta di regressione dei minimi quadrati.
Le soluzioni possno esser reperite facilmente considerando la soluzione del seguente sistema di 2 equazioni (dette equazioni normali):
\[\begin{align} \frac{\partial L(\beta_0,\beta_1}{\partial \beta_0} &= -2\sum_{i=1}^n (y_i-\beta_0-\beta_1 x_i)=0\\ \frac{\partial L(\beta_0,\beta_1}{\partial \beta_1} &= -2 \sum_{i=1}^n (y_i-\beta_0-\beta_1 x_i)x_i= 0 \end{align}\]
La soluzione del sistema, dopo alcuni passaggi, conduce ai seguenti valori per \(\hat{\beta_0}\) e \(\hat{\beta_1}\) che rappresentano quindi le soluzioni dei minimi quadrati:
\[\hat{\beta_0}=M(y)-\hat{\beta_1}M(x)\]
\[\hat{\beta_1}=\frac{cov(XY)}{var(X)}\] Ove \(M(y)\) e \(M(x)\) denotano le medie calcolate per i valori osservati delle due variabili \(Y\) e \(X\).
Nel caso siano coinvolte solo due variabili si parla di regressione lineare semplice.
Di solito l’interesse maggiore è sul coefficiente angolare della retta \(\hat{\beta_1}\) che è detto coefficiente di regressione. Se esso fosse nullo la retta è parallela all’asse orizzontale e le medie condizionate \(M(Y|x)\) sarebbero tutte uguali tra loro. Non c’è quindi dipendenza lineare fra le variabili: si noti che in effetti il coefficiente di regressione è 0 se la covarianza è zero.
Si noti anche che il coefficiente di regressione angolare può anche essere scritto in funzione del coefficiente di correlazione fra le due variabili come segue \[\hat{\beta_1}=r(X,Y)\frac{sd(Y)}{sd(X)}\].
Si possono quindi calcolare i valori sulla retta dei minimi quadrati \(\hat{y}_i=\hat{\beta_0}+\hat{\beta_1}x_i\) e quindi i residui che corrispondono alla soluzione dei minimi quadrati \(y_i-\hat{y}_i= r_i\) e ottenere, infine, la somma dei quadrati dei residui \(SQE=\sum_{i=1}^n(y_i-\hat{y}_i)^2\).
Si noti peraltro che in virtù della prima equazione normale è \(\sum_{i=1}^n(y_i-\hat{y}_i)=0\).
# proviamo per i dati whiteside a ottenere la retta di regressione dei minimi quadrati
dev=sum((Temp-mean(Temp))^2)
b1<- cov(Gas,Temp)/var(Temp)
b1
print(paste("coefficiente di regressione",b1))
b0<- mean(Gas)-b1*mean(Temp)
b0
print(paste("intercetta",b0))
# otteniamo anche i valori sulla retta e la somma dei quadrati dei residui
Gasprev<-b0+b1*Temp
res<-Gas-Gasprev
SQE=sum(res^2)
print(paste("Devianza dei residui",SQE))
# e aggiungiamo la retta al diagramma di dispersione
plot(Temp,Gas, pch=19)
abline(b0,b1, col=2)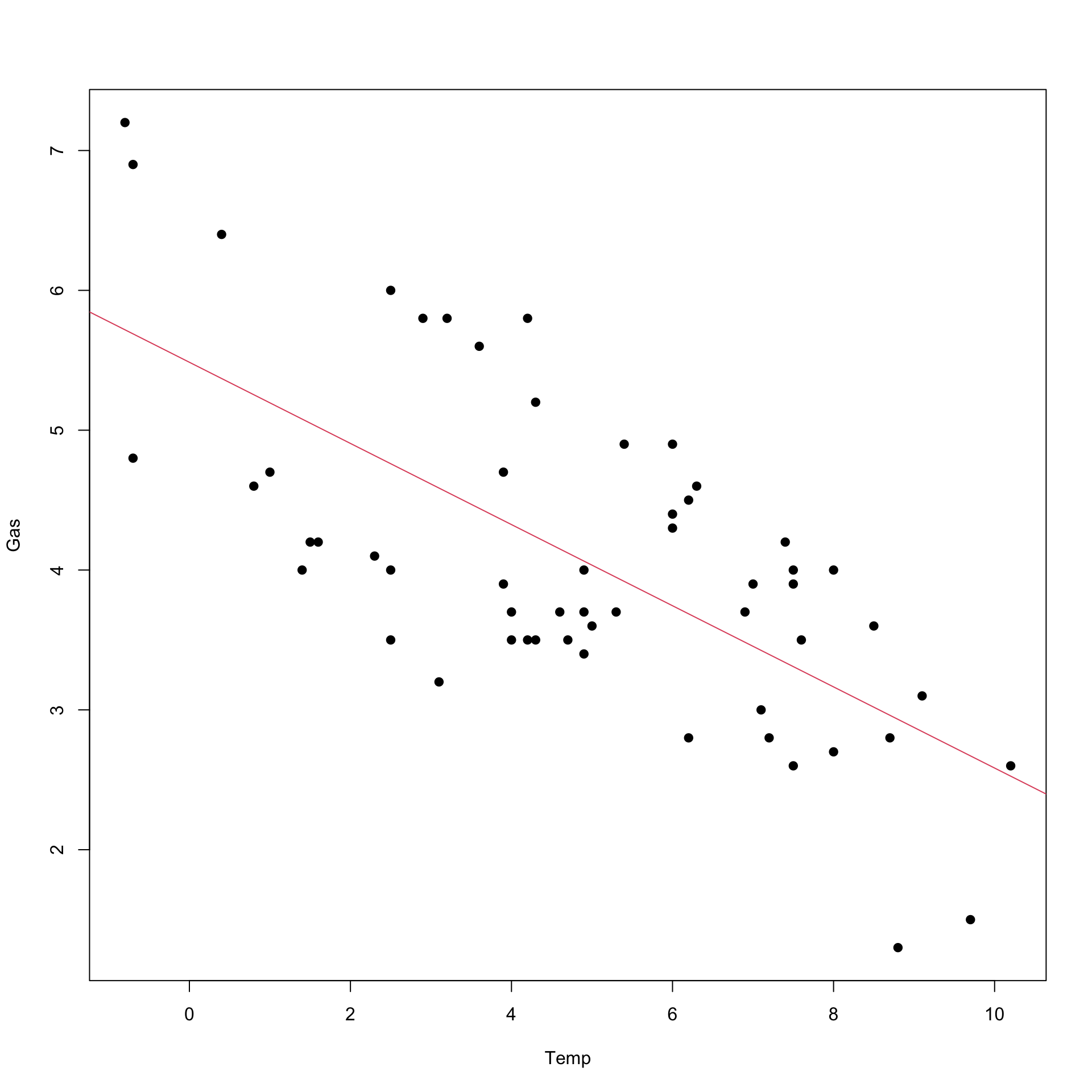
## [1] -0.2902082
## [1] "coefficiente di regressione -0.290208150455141"
## [1] 5.486193
## [1] "intercetta 5.48619330489738"
## [1] "Devianza dei residui 39.9948681988638"
##
## Call:
## lm(formula = Gas ~ Temp)
##
## Coefficients:
## (Intercept) Temp
## 5.4862 -0.2902Si potrebbe cercare una approssimazione dei punti con una retta scelta secondo altri criteri che potrebbero essere sensati, ma in tal caso se si decide che la misura della qualità dell’approssimazione fornita dalla retta sia misurata dalla somma dei quadrati delle distanze dei punti dalla retta (SSE), tale misura risulterà più elevata.
Vediamo un esempio. Si ottenga una retta che approssima i punti con un altro criterio. Consideriamo la retta che passa fra i rispettivi quartili delle due variabili e chiediamo poi che essa passi per il punto di coordinate \((M(y),M(x))\).
# otteniamo la retta che passa per i quartili per i dati visti sopra
b1q<- -(quantile(Gas,0.75)-quantile(Gas,0.25))/(quantile(Temp,0.75)
-quantile(Temp,0.25))
# imponiamo che passi per i punti medi
b0q<- mean(Gas)-b1q*mean(Temp)
plot(Temp,Gas, pch=19)
abline(b0,b1, col=2)
abline(b0q,b1q,col=3)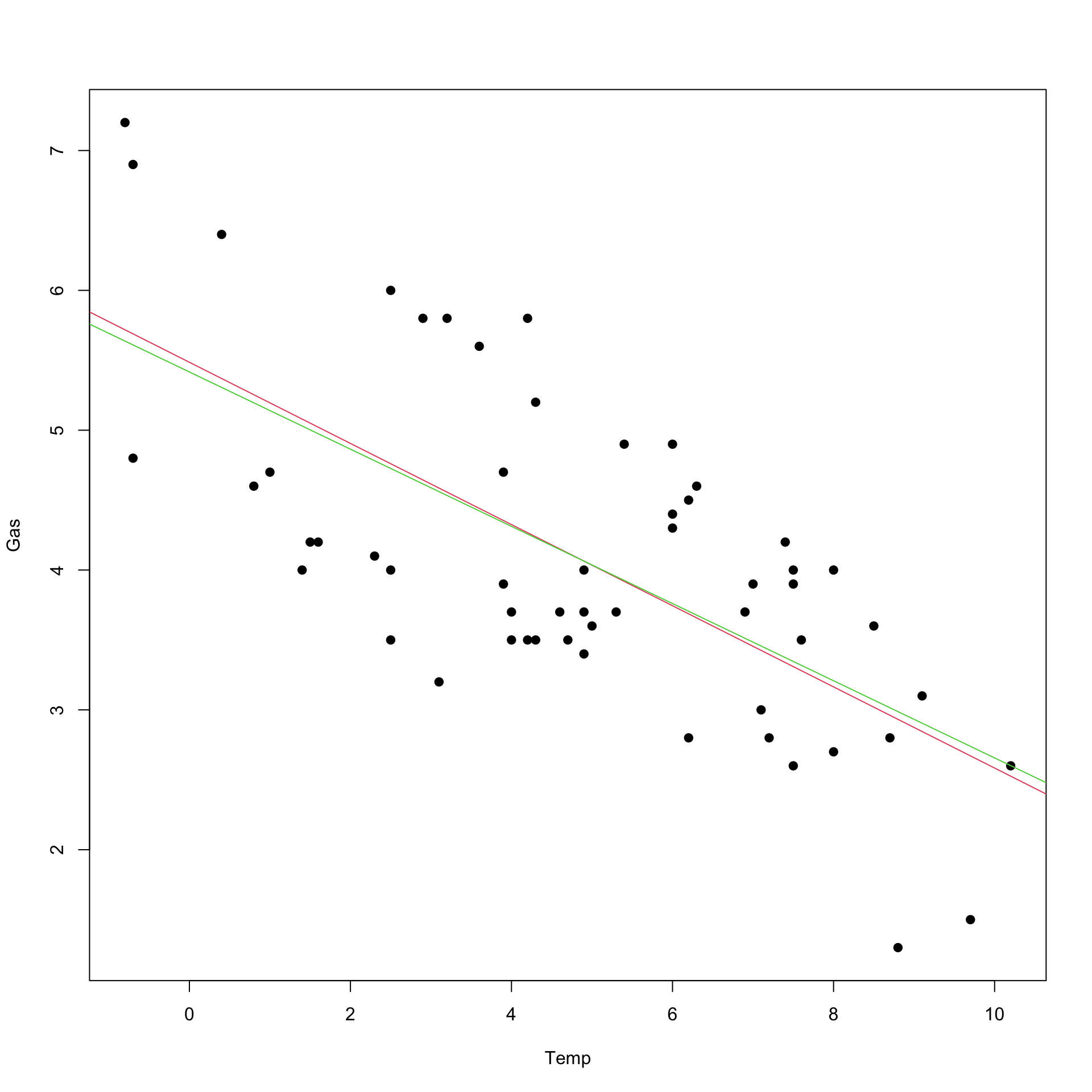
# aggiungiamo anche tale retta al diagramma di dispersione
# otteniamo i valori su tale retta e calcoliamo
# la somma dei quadrati dei residui
Gasprevq<-b0q+b1q*Temp
resq<-Gas-Gasprevq
SQE2=sum(resq^2)
print(paste("Devianza dei residui",SQE2))
# si noti che è più grande di quello già ottenuto con i minimi quadrati## [1] "Devianza dei residui 40.077939784819"5.3.3.2 Misurare la qualità della regressione: il coefficiente di determinazione lineare
La variabilità complessiva della quantità di interesse \(Y\) se non si disponesse (o se non si utilizza) anche l’informazione su \(x\) è misurata dalla devienza \[\sum_{i=1}^n (y_i - \overline{y})^2 \qquad \mbox{Somma dei quadrati dei residui totale (SQT)}\] Si noti che questa è una misura dell’incertezza su \(Y\) se utilizzassimo come sintesi della variabile la sua media.
- La devianza totale SQT di \(Y\) dopo aver considerato la regressione dei minimi quadrati, può essere scomposta come segue:
devianza di regressione SQR che è la parte di variabilità di \(Y\) spiegata dal modello di regressione: \[\sum_{i=1}^n (\hat{y}_i - \overline{y})^2 \qquad \mbox{devianza di regressione SQR}\]
devianza dei residui SQE che rappresenta la variabilità dell’errore che commettiamo utilizzando \(\hat{y}_1\): \[\sum_{i=1}^n (y_ i - \hat{y}_i)^2 \qquad \mbox{variabilità dell'errore (devianza dei residui) SQE}\]
- Si può verificare facilmente che per la regressione dei minimi quadrati: \[ \underset{\scriptsize{\bf{\mbox{SST}}}}{\sum_{i=1}^n (y_i - \overline{y})^2} = \underset{\scriptsize{\bf{\mbox{SSE}}}}{\sum_{i=1}^n ( y_i - \hat{y_i})^2} + \underset{\scriptsize{\bf{\mbox{SSR}}} }{\sum_{i=1}^n (\hat{y}_i - \overline{y})^2}\]
Si noti che tale scomposizione è parallela a quella già discussa nel caso nell’analisi di una variabile quantitativa analizzata per gruppi.
Se la retta di regressione interpreta bene i dati allora:
- SSE, devianza dei residui, è piccola rispetto alla SST (devianza totale)
- SSR, la devianza spiegata dalla regressione, è la parte principale di SST
Possiamo quindi introdurre il seguente coefficiente di determinazione (lineare): \[R^2 = \frac{\mbox{SSR}}{\mbox{SST}} = 1- \frac{\mbox{SSE}}{\mbox{SST}} \]
- \(0 \leq R^2 \leq 1\) e indica la quota di variabilità che è spiegata dalla regressione
- Più basso è \(R^2\) peggiore è l’adattamento del modello ai dati
- Se fosse pari a 1 indicherebbe un perfetto allineamento dei dati
- Si può infine dimostrare che \(R^2=r(X,Y)^2\), cioè è pari al quadrato del coefficiente di correlazione lineare fra le due variabili.
Si noti tuttavia che se il coefficiente di correlazione lineare, e di conseguenza il coefficiente di determinazione lineare, è da ritenersi nullo (molto vicino a 0), concluderemo che non c’è relazione lineare fra le variabili.
Attenzione: questo non significa che possa esserci una relazione fra le variabili di natura diversa. Ad esempio, i punti nel diagramma di dispersione potrebbero disporsi lungo una parabola e suggerire la presenza di una relazione fra le variabili non di tipo lineare. Quindi l’assenza di relazione lineare non implica l’indipendenza delle variabili.
5.3.3.3 la funzione lm() e il modello lineare
Dato il rilievo che ha l’analisi di regressione lineare non è sorprendente che in R vi sia una funzione che svolge tale analisi. Tale funzione, che come si vedrà può essere generalizzabile al caso dell’analisi con più variabili, fornisce tutte le informazioni rilevanti.
Tuttavia prima di introdurre la funzione lm() e per comprendere al meglio alcuni suoi risultati occorre introdurre il modello di regressione lineare.
Il modello di regressione lineare (lm sta appunto per linear model) è un modello statistico che è formulato per l’ambito inferenziale: i dati osservati sono visti come determinazioni di variabili aleatorie sulle quali si fanno alcune specifiche assunzioni. Come in ogni modello statistico vi sono dei parametri ignoti che caratterizzano le variabili aleatorie e su questi parametri si fa inferenza (secondo le procedure di stima, intervalli di confidenza, verifica di ipotesi) avendo osservato un campione casuale di dati.
Il modello lineare ha la seguente specificazione: \[Y_i=\beta_0+\beta_1x_i+\epsilon_i\] \(\epsilon_i\) sono determinazioni di una variabile aleatoria sulle quali si fanno le seguenti assunzioni: - \(E(\epsilon_i)=0\) - \(Var(\epsilon_i)=\sigma^2\) (omoschedasticità ovvero stessa varianza \(\forall i\)). - \(E(\epsilon_i,\epsilon_j)=0~~~\forall i\neq j\) ovvero i residui sono incorrelati.
Inoltre è molto comune fare l’ulteriore assunzione di gaussianità per \(\epsilon_i\), cioè \(\epsilon_i\sim \mathbb{N}(0,\sigma^2)\)
Con le seguenti assunzioni quindi i dati osservati \(Y_i \sim \mathbb{N}(\beta_0+\beta_1x_i,\sigma^2)\) sono determinazioni di gaussiane la cui media varia con \(x\) e i dati \(x_i\) sono non stocastici.
La determinazione dei parametri del modello (\(\beta_0,\beta_1, \sigma^2)\), usando i metodi dell’inferenza statistica, può essere affrontata in vari modi. Si osservi però che se si accetta l’assunzione di normalità della componente aleatoria \(\epsilon_i\) il metodo più adeguato per ottenere tali parametri è ancora il criterio dei minimi quadrati già introdotto. Inoltre la varianza dei residui \(\hat{\sigma}^2\) viene ottenuta considerando la somma dei quadrati dei residui divisa per \(n-2\) cioè \(\hat{\sigma}^2=SQE/(n-2)\).
In ambito inferenziale, come discusso per i precedenti esempi si può valutare se il coefficiente angolare della retta \(\hat{\beta}_1\) è abbastanza piccolo così da ritenere che sia nullo e che lo scostamento rispetto a 0 è da ritenersi dovuto solo al caso. Se, ad esempio, possiamo ritenere che \(\beta_1\) è in effetti pari a 0 allora non vi è relazione lineare e \(X\) non è utile a prevedere \(Y\) secondo questo modello. Tuttavia non possiamo semplicemente guardare al valore di \(\beta_1\) perchè il suo ordine di grandezza dipende dalla scala di misura delle due variabili coinvolte e dalla qualità della rappresentazione della relazione lineare (ovvero da quanto i punti si discostano dalla retta di regressione).
Tuttavia, anche in tal caso si può associare al valore di \(\hat{\beta}_1\) ottenuto a partire dai dati, una probabilità, il \(p-value\), che è in relazione a quanto si può ritenere plausibile osservare tale valore (o valori ancora più estremi cioè relativi a casi in cui il coefficiente angolare è ancora più distante da 0) se in effetti non vi fosse alcuna relazione lineare, cioè se \(\beta_1=0\). In questo caso, si può calcolare \(\frac{\hat{\beta}_1}{\sqrt{\hat{\sigma}^2_{(\beta_1)}}}\).
La quantità al denominatore dell’ultima espressione, che dipende da \(SQT\) e dalla varianza di \(X\), consente di standardizzare il valore di \(\beta_1\) e di fare riferimento a una particolare distribuzione di probabilità (che verrà introdotta nei corsi di inferenza statistica ed è denominata t di student). Con riferimento ad essa si potrà calcolere il \(p-value\). E, ancora una volta, quanto più questo valore è prossimo a 0 tanto più i dati non danno sostegno all’ipotesi che non esista una relazione lineare fra le variabili.
Illustriamo quindi l’uso della funzione lm() e i dei risultati principali che fornisce usando ancora i dati whiteside.
5.3.4 Il lisciamento della curva di regressione
Assumere che le medie condizionate \(M(Y|x)\) siano allineate lungo una retta (o un’altra curva semplice come la parabola) rende agevole l’analisi ma i dati potrebbero suggerire un comportamento molto diverso. Un approccio alternativo è quindi quello di esplorare metodi che consentano di tracciare la curva di regressione liberamente lasciando che siano i dati stessi a suggerire la forma della curva.
Un semplice espediente potrebbe esser quello di suddividere la variabile \(X\) in classi e calcolare la media dei valori \(y_i\) per i dati che sono in quella classe. A quel punto si potrebbe calcolare la media condizionata e porla costante all’interno di ogni classe. Questo porterebbe a una funzione costante a tratti la cui forma è suggerita dai dati.
Tuttavia sarebbe desiderabile, anche per una visualizzazione dei dati sintetica, che la funzione di regressione sia liscia senza sbalzi. In tal senso il problema è simile a quello affrontato nel caso del lisciamento della funzione di densità come alternativa all’istogramma.
La funzione di regressione descrive il comportamento della media della variabile dipendente condizionata ai valori in corrispondenza dei valori di una variabile esplicativa. Anche se l’idea che tali medie stiano lungo funzioni semplici come la retta è spesso utile, se guardiamo al diagramma di dispersione si ha spesso l’impressione che le cose possano essere più complicate. Potrebbe rivelarsi interessante che i dati stessi ci suggeriscano l’andamento delle medie di \(Y\) condizionatamente ai valori di \(x\).
Il grafico che segue contiene la nuvola di punti relativa a due variabili: il prezzo delle case vendute in un certo periodo ad Albuquerque (New Mexico), \(y\), e la loro dimensione (in piedi quadrati), \(x\). Che fra le due variabili ci sia una relazione è scontato, che il prezzo medio delle case vari linearmente al variare della dimensione lo è un po’ meno.
detach(whiteside)
# leggiamo i dati dal file case.csv
case<-read.csv("case.csv")
plot(case$SquareFeet,case$Price)
modl<-lm(Price~SquareFeet, data=case) # otteniamo i parametri di un modello lineare
abline(modl, col=2)
# usiamo la funzione ksmooth per ottenere un lisciamento col metodo del nucleo
# la funzione vuole le due variabili inserite come in plot
# ed è specificato il parametro baandwidth che regola il lisciamento
lines(ksmooth(case$SquareFeet,case$Price, bandwidth=1400), col=4, lty=2)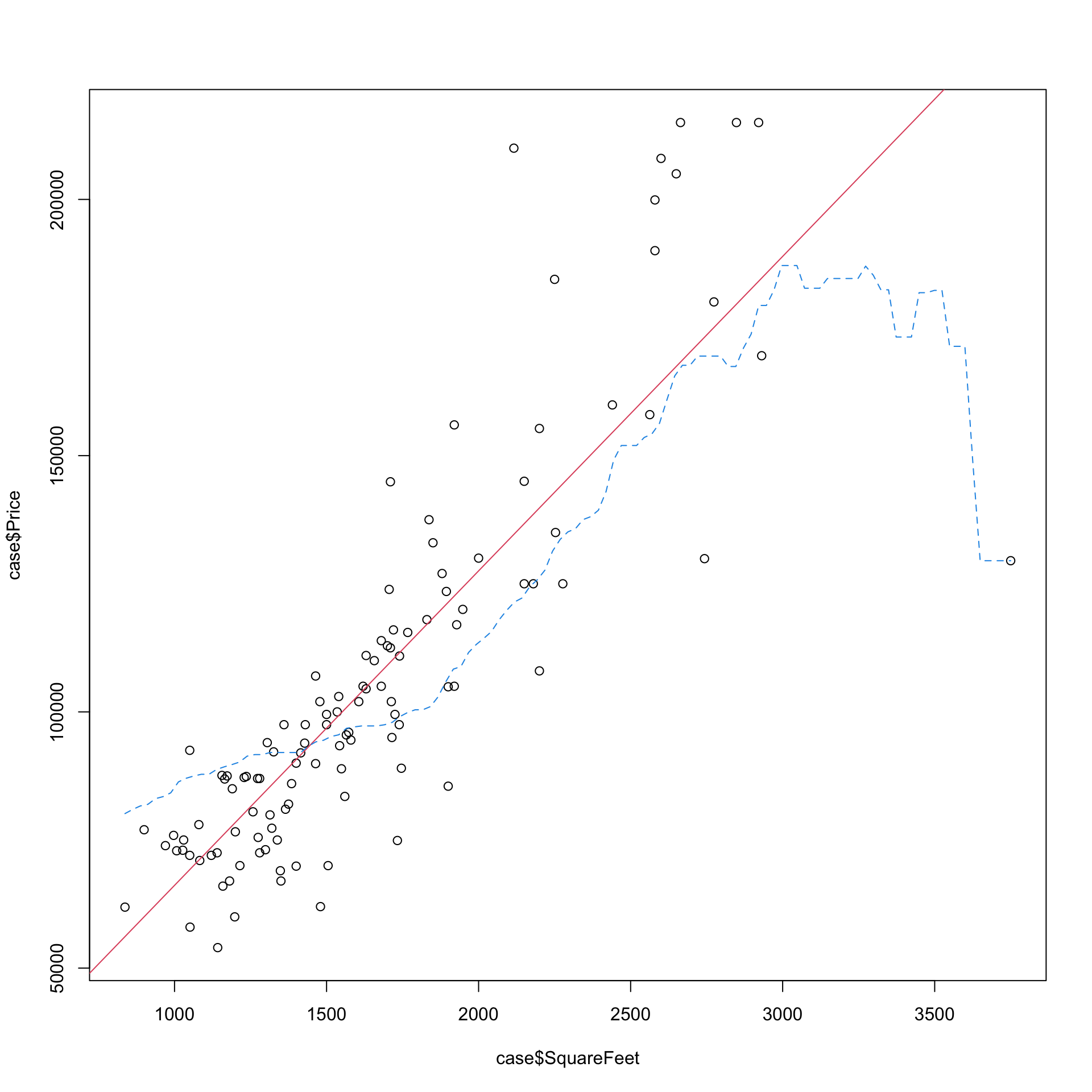
Nel grafico è riportata la retta dei minimi quadrati e una curva (più o meno) liscia che tende a seguire con meno rigidità l’andamento della nuvola; peraltro, ci si potrebbe, ad esempio, ragionevolmente attendere che aumenti della metratura per case molto grandi si tradurranno in aumenti di prezzo inferiori a quelli che si osservano per le case piccole (ci si aspetta che la derivata decresca). In realtà, per i dati americani questo sembra non succedere.
La funzione che passa tra i punti è una versione della curva di regressione ottenuta non parametricamente con il metodo del nucleo. In particolare si è utilizzato il cosidetto metodo delle medie locali. Esso consiste nel calcolare in corrispondenza di ciascun valore \(x\) (sia esso osservato o meno) la media delle coordinate \(y\) per le unità che sono vicine ad \(x\) (si calcola cioè una media locale). Questo si potrebbe realizzare prendendo una “striscia”, più o meno stretta, attorno ad \(x\) e considerando la media delle coordinate \(y\) solo per le unità nella striscia. Si può tuttavia fare di meglio: per calcolare la funzione \(m(x_0)=Media(y|x_0)\) in corrispondenza di un dato valore \(x_0\), si possono considerare tutte le unità statistiche e fare una media ponderata di tutti i valori \(y\) dando però pesi maggiori a quelle unità che hanno valori di \(x\) vicini a \(x_0\).
La funzione che determina questi pesi è una funzione nucleo \(K()\) che ha le stesse caratteristiche viste nel caso della determinazione della funzione di densità. Si ottiene la seguente media ponderata \[\begin{equation} m(x)= \frac{\sum_{i=1}^{n} y_i K\left(\frac{x-x_i}{h}\right)}{\sum_{i=1}^{n}K\left(\frac{x-x_i}{h}\right)}. \end{equation}\].
Si tratta quindi ancora del metodo del nucleo, di solito \(K()\) è una normale standard, e ancora una volta è determinante il valore di h: se troppo piccolo accade che la curva tende a seguire troppo da vicino ciascun punto ovvero adattarsi troppo ad ogni particolarità dei dati, se troppo grande praticamente tutte le osservazioni hanno lo stesso peso per ogni \(x\) e quindi si ottiene semplicemente una retta parallela all’asse \(X\) all’altezza della media di \(Y\). Questo comportamente è un esempio del cosiddetto trade-off tra bias e varianza: ovvero fra una funzione liscia che ha molto bias, cioè rischia di essere diversa dalla funzione che misura la relazione far le variabili, e una molto accidentata che è molto variabile.
Nel caso della regressione si può fare ancora di meglio: ad esempio, si possono usare polinomi locali invece che medie locali, oppure si può tenere conto della densità locale dei punti \(x\) o, ancora, utilizzare metodi più resistenti (che risentono meno di valori anomali). Un metodo che fa tutto questo è il metodo “Lowess” (per il quale esiste la funzione lowess() in R).
Per avere solo un’idea di cosa verrebbe fuori utilizzando il Lowess si veda la figura sopra (si noti come il valore a destra nelle figura abbia, con il Lowess, meno rilevanza nel determinare la funzione).
# leggiamo i dati dal file case.csv
case<-read.csv("case.csv")
plot(case$SquareFeet,case$Price)
modl<-lm(Price~SquareFeet, data=case) # otteniamo i parametri di un modello lineare
abline(modl, col=2)
# usiamo ora la funzione lowess per ottenere un lisciamento col metodo dei
# polinomi locali in una versione resistente ai valori anomali
#
lines(lowess(case$Price~case$SquareFeet, f=.8), col=5, lwd=2)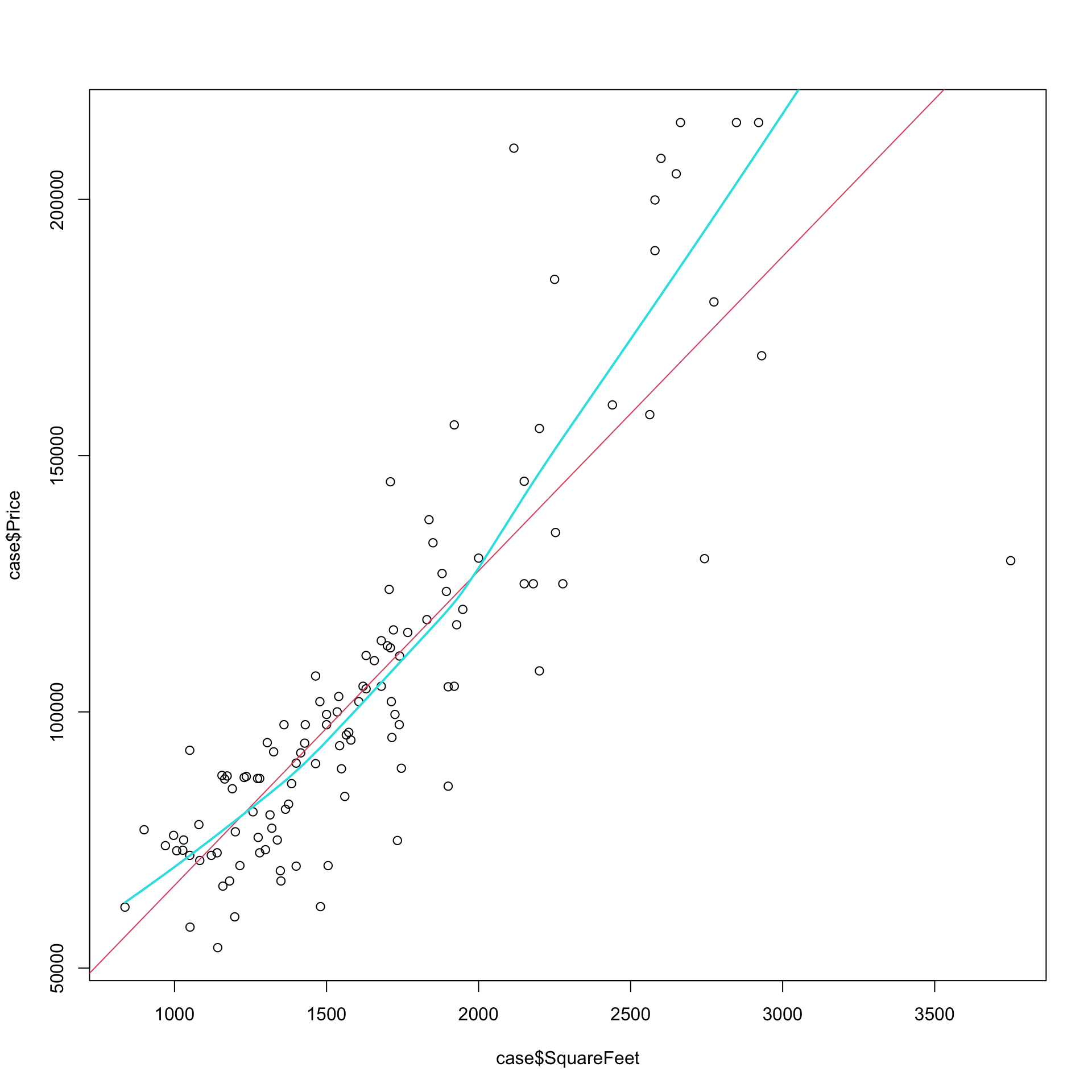
Esistono altre funzioni ad esempio nel pacchetto KernSmooth per ottenere la funzione con il metodo del nucleo.
Si noti che il lisciamento con il metodo del nucleo risulta più robusto in quanto include automaticamente un metodo per dare meno peso a valori anomali (ad esempio la curva risente meno del punto a destra).
L’uso di versioni lisciate della curva di regressione al fine di visualizzare i dati su due variabili esplicative è diventato ormai consueto.
Pertanto non stupisce che nei sistemi di visualizzazione, come ad esempio ggplot2, sia molto semplice ottenere le curve tipo lowess (o loess).
library(ggplot2)
ggplot(mapping = aes(x = SquareFeet, y = Price), data = case) +
geom_point() +
geom_smooth(aes(), se=F)## `geom_smooth()` using method = 'loess' and formula = 'y ~ x'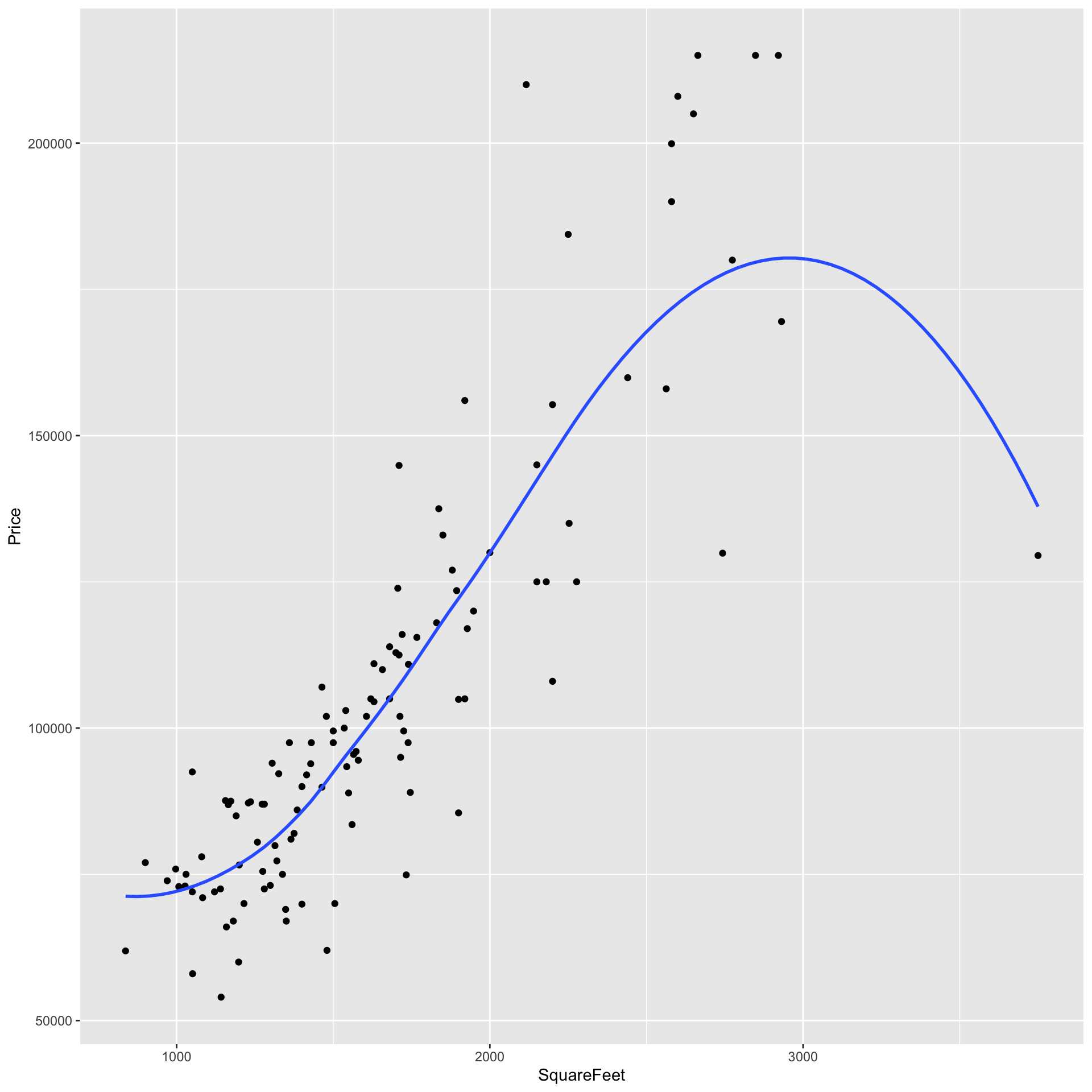
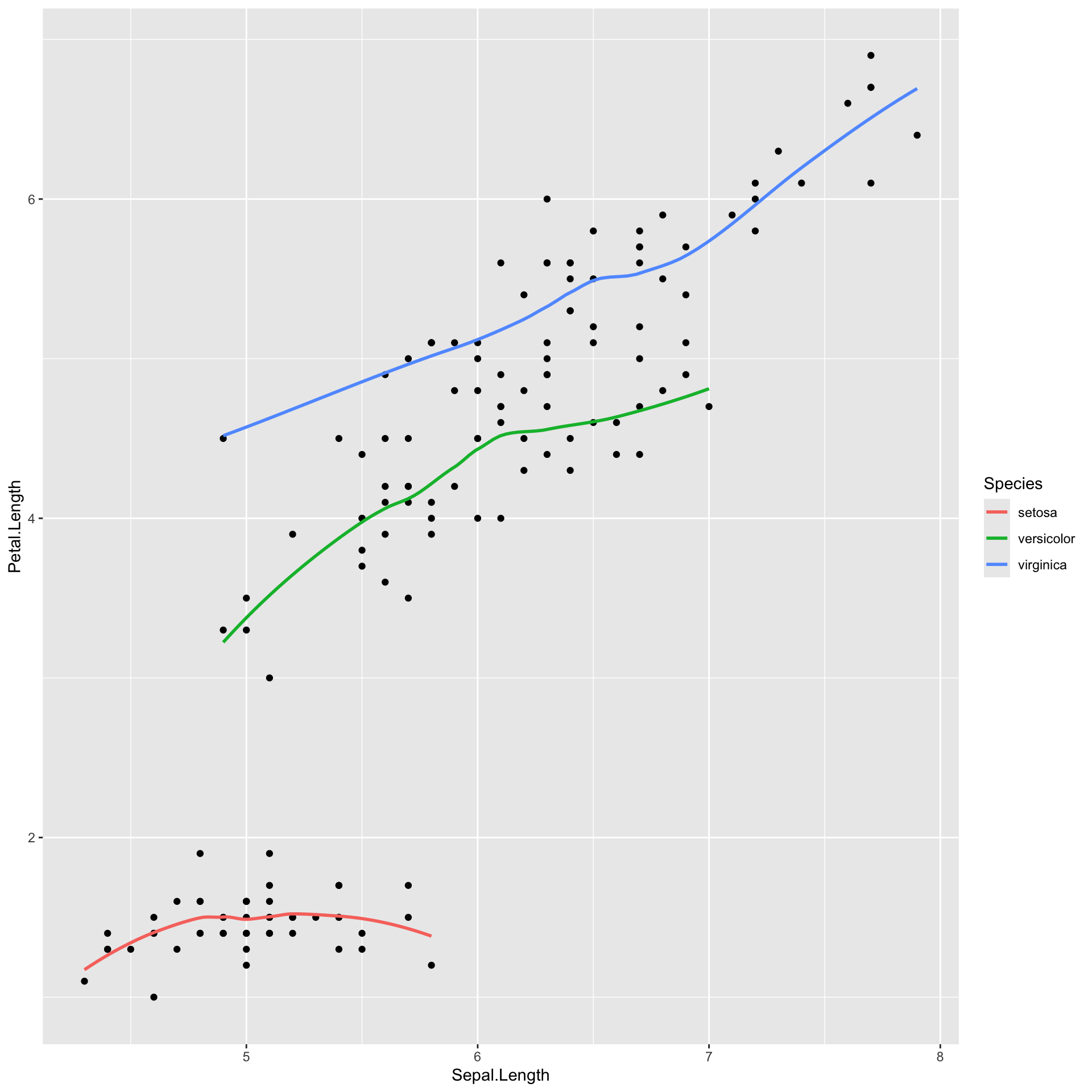
Sotto si riporta anche un esempio con i dati iris in cui le curve loess sono mostrate per sottogruppi di dati.
ggplot(mapping = aes(x = Sepal.Length, y = Petal.Length), data = iris) +
geom_point() +
geom_smooth(aes(colour=Species), se=F)## `geom_smooth()` using method = 'loess' and formula = 'y ~ x'